El trabajo en ecología y cualquiera de sus disciplinas
requiere información actualizada, o al menos estandarizada, sobre la
nomenclatura de nuestros organismos de estudio (Patterson
et al. 2010). Para solventar este problema, se han ido desarrollando
diferentes herramientas que permitan cotejar la nomenclatura de una especie
contra bases de datos taxonómicas. Por un lado, tenemos herramientas como las
webs de Flora Ibérica y Fauna Ibérica, que permiten
consultar de forma manual sinónimos y nombres. Sin embargo, estas carecen de
una interfaz de usuario que permita la consulta de un gran número de especies,
acceder a ellas de forma automática o hacerlo desde entornos que permitan
integrar los resultados en un flujo de trabajo más complejo. Por otro lado,
existen herramientas que sí pueden consultarse a través de interfaces de
programación de aplicaciones (API) desde entornos como R (R
Core Team 2025), facilitando la tarea de cotejar un gran número de especies
a la vez, así como su incorporación en nuestros flujos de trabajo. Algunos
ejemplos de esto son el paquete taxize (Chamberlain
et al. 2020), con acceso a más de 20 bases de datos diferentes; o el
paquete WorldFlora (Kindt 2020) para acceder a World
Flora Online. La principal desventaja de estos servicios radica en que, a
pesar de la gran cantidad de información que contienen, puede darse un
desajuste entre su nomenclatura y la establecida a nivel nacional o regional.
Esta limitación se hace más patente si, por ejemplo, queremos recopilar
información sobre el estado de conservación de una especie presente en España,
puesto que el nombre aceptado en una base de datos internacional puede no casar
con el empleado en catálogos a nivel nacional o regional.
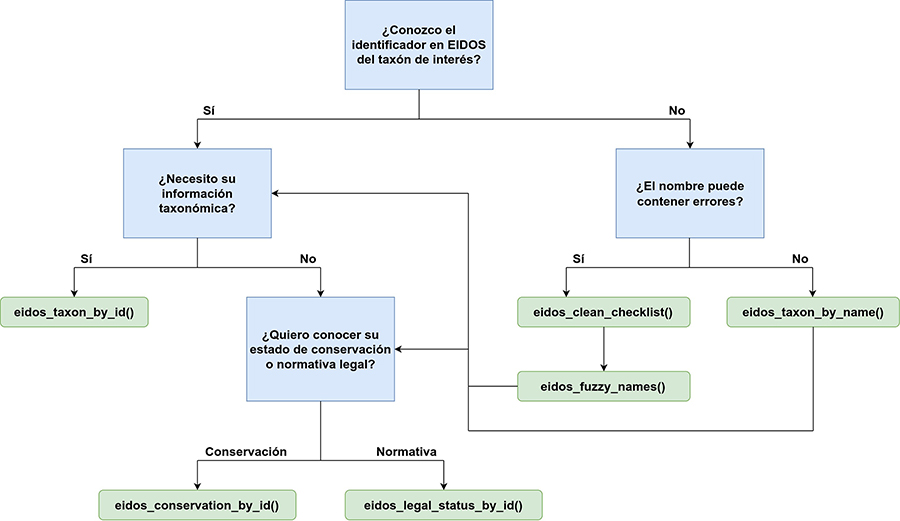
Figura 1. Diagrama del flujo de trabajo básico
con eidosapi.
Figure
1. Basic workflow with eidosapi.
Instalación y ejemplos básicos de uso de eidosapi
La instalación del paquete puede realizarse fácilmente
clonando el repositorio disponible en GitHub, donde también se podrán encontrar
las nuevas actualizaciones.
remotes::install_github("https://github.com/hmirceb/eidosapi",
force = TRUE,
quiet = TRUE)
library(eidosapi)
Búsqueda de especies por
nombre
Un ejemplo básico sería buscar el nombre aceptado y
sinónimos de una o varias especies. Para ello vamos a emplear el halcón de
Eleonora (Falco eleonorae), que no tiene sinónimos; y Polygonum
viviparum que tiene varios. El procedimiento básico consiste en crear una
tabla con el género y la especie de cada taxón. También podemos incluir una
columna con la subespecie y la autoridad taxonómica que haya descrito el taxón,
en este caso vamos a probar con Androsace cylindrica hirtella. En el
caso de que el taxón que nos interesa no tuviese subespecies o no conociésemos
la autoría podemos omitir las columnas correspondientes o rellenarlas con NA.
Cabe destacar que el IEPNB todavía es un trabajo en marcha y algunos organismos
no están presentes en la base de datos o carecen de información detallada. En
esos casos eidosapi devuelve una fila vacía en la tabla final pero
indicando el nombre que hayamos buscado.
taxa_list <- data.frame(
genus = c("Falco", "Polygonum", "Androsace"),
species = c("eleonorae", "viviparum", "cylindrica"),
subspecies = c(NA, NA, "hirtella")
)
eidos_results <- eidos_taxon_by_name(
taxa_list =
taxa_list
)
knitr::kable(eidos_results[c("supplied_taxon",
"idtaxon", "nametype", "acceptednameid")])
|
supplied_taxon
|
idtaxon
|
nametype
|
acceptednameid
|
|
Falco eleonorae
|
11198
|
Aceptado/válido
|
11198
|
|
Polygonum viviparum
|
32824
|
Sinónimo
|
7277
|
|
Androsace cylindrica
hirtella
|
2146
|
Aceptado/válido
|
2146
|
Estado de conservación
La columna idtaxon obtenida en el paso anterior
contiene el identificador único para cada taxón en el IEPNB. Si quisiéramos
saber si el halcón de Eleonora tiene asociada alguna categoría de amenaza, solo
tendríamos que introducir este indicador en la función eidos_conservation_by_id().
eidos_results <- eidos_taxon_by_name(
taxa_list = "Falco
eleonorae"
)
print(eidos_results$idtaxon)
[1] "11198"
eidos_cons <- eidos_conservation_by_id(
taxon_id =
eidos_results$idtaxon
)
knitr::kable(eidos_cons[c("name_clean", "idtaxon", "anio", "categoriaconservacion", "aplicaa")])
|
name_clean
|
idtaxon
|
anio
|
categoriaconservacion
|
aplicaa
|
|
Falco eleonorae
|
11198
|
1992
|
R (Rara)
|
España
|
|
Falco eleonorae
|
11198
|
2004
|
NT (Casi amenazado)
|
España
|
|
Falco eleonorae
|
11198
|
2016
|
LC (Preocupación
menor)
|
Mundial
|
|
Falco eleonorae
|
11198
|
2017
|
LC (Preocupación
menor)
|
Mundial
|
|
Falco eleonorae
|
11198
|
2019
|
LC (Preocupación
menor)
|
Mundial
|
|
Falco eleonorae
|
11198
|
2021
|
LC (Preocupación
menor)
|
Mundial
|
|
Falco eleonorae
|
11198
|
2021
|
NT (Casi amenazado)
|
Península
|
En este caso, el halcón de
Eleonora a nivel mundial está en la categoría “Preocupación menor” (LC) desde
el año 2016, mientras que en España y la Península Ibérica se la considera
“Casi amenazado” (NT) desde 2004 y 2021 respectivamente. Siguiendo este mismo
procedimiento podríamos conocer las normativas específicas que rigen esas
categorías para cada especie con la función eidos_legal_status_by_id().
Búsqueda de especies con errores en la nomenclatura
Un problema común a la hora de trabajar con datos de
especies son los errores de escritura. El paquete eidosapi incluye la
función eidos_fuzzy_names() que permite consultar en la
LP, previa descarga con la función eidos_clean_checklist(),
los nombres que más se acerquen a la información que hayamos aportado. Podemos
comprobarlo con algunos nombres muy mal escritos.
taxa_list <- data.frame(
genus = c("Vorderea", "Alytes"),
species = c("pyrenaica", "cisternasi")
)
eidos_result <- eidos_fuzzy_names(taxa_list =
taxa_list)
Downloading checklist and formatting,
please wait...
knitr::kable(eidos_result[c("supplied_taxon", "idtaxon",
"name", "withoutautorship")])
|
supplied_taxon
|
idtaxon
|
name
|
withoutautorship
|
|
Vorderea pyrenaica
|
94856
|
Borderea pyrenaica
Miégev.
|
Dioscorea pyrenaica
|
|
Alytes cisternasi
|
10909
|
Alytes cisternasii
Boscá, 1879
|
Alytes cisternasii
|
La función eidos_fuzzy_names()
también nos permite ajustar la búsqueda empleando el argumento maxdist,
que controla el grado de similitud entre los nombres aceptados y el nombre que
queremos buscar en una escala de 0 (indicando una coincidencia exacta) a 1.
taxa_list <- data.frame(
genus = c("Nepeta", "Barlia"),
species = c("beltrani", "robertianum")
)
checklist <- eidos_clean_checklist()
fuzzy_dist2 <- eidos_fuzzy_names(taxa_list =
taxa_list,
checklist =
checklist,
maxdist = 0.05)
knitr::kable(fuzzy_dist2[c("supplied_taxon", "idtaxon",
"name", "withoutautorship")])
|
supplied_taxon
|
idtaxon
|
name
|
withoutautorship
|
|
Nepeta beltrani
|
6550
|
Nepeta beltranii Pau
|
Nepeta hispanica
|
|
Barlia robertianum
|
NA
|
NA
|
NA
|
fuzzy_dist3 <- eidos_fuzzy_names(taxa_list = taxa_list,
checklist
= checklist,
maxdist
= 0.1)
knitr::kable(fuzzy_dist3[c("supplied_taxon", "idtaxon",
"name", "withoutautorship")])
|
supplied_taxon
|
idtaxon
|
name
|
withoutautorship
|
|
Nepeta beltrani
|
6550
|
Nepeta beltranii Pau
|
Nepeta hispanica
|
|
Barlia robertianum
|
32238
|
Barlia robertiana
(Loisel.) Greuter
|
Himantoglossum
robertianum
|
Conclusiones
El paquete eidosapi permite acceder de forma sencilla
a las bases de datos taxonómicas del Inventario del Patrimonio Natural y de la
Biodiversidad, facilitando la estandarización de nombres científicos, obtener
información detallada sobre sobre su estado de conservación y permitiéndonos
incorporar esta información a otros flujos de trabajo.
Disponibilidad de datos y código
El código del paquete eidosapi está disponible en
GitHub (https://github.com/hmirceb/eidosapi)
y Zenodo (Hmirceb 2025; https://doi.org/10.5281/zenodo.17505969).
Financiación, permisos requeridos, potenciales
conflictos de interés y agradecimientos
Querría mostrar mi agradecimiento a Julen Astigarraga,
Ignacio Ramos Gutiérrez e Ignasi Bartomeus por sus comentarios y sugerencias
para mejorar tanto esta nota como el paquete eidosapi. Esta nota se ha
revisado siguiendo un proceso colaborativo y público disponible en https://github.com/ecoinfAEET/Notas_Ecosistemas/issues/61.
El autor declara no tener conflicto de intereses.
Referencias
Chamberlain, S., Szoecs, E., Foster, Z., Arendsee, Z., Boettiger, C., Ram, K.,
Bartomeus, I., et al. 2020. taxize: Taxonomic information from around the web.
Hmirceb 2025. hmirceb/eidosapi: eidosapi 1.1.0 (v1.1.0). Zenodo.
Kindt, R. 2020. WorldFlora: An R package for exact and fuzzy matching of
plant names against the World Flora Online taxonomic backbone data. Applications
in Plant Sciences 8: e11388.
Ooms, J. 2014. The jsonlite Package: A Practical and Consistent Mapping Between
JSON Data and R Objects. arXiv:1403.2805 [stat.CO].
Patterson, D.J., Cooper, J., Kirk, P.M., Pyle, R.L., Remsen, D.P. 2010. Names are
key to the big new biology. Trends in Ecology & Evolution
25: 686-691.
R Core Team 2025. R: A
Language and Environment for Statistical Computing. R Foundation
for Statistical Computing, Vienna, Austria.
Robinson, D. 2025. fuzzyjoin: Join Tables Together on Inexact Matching.
