Inteligencia artificial en
ecología, una pequeña introducción para principiantes
La inteligencia artificial (IA) está transformando
rápidamente muchas áreas del conocimiento, y la ecología no es una excepción (p. ej., Borowiec et al. 2022;
Pichler y Hartig 2023;
Wilson 2024). Con el aumento de datos ambientales y la
necesidad de análisis complejos, la IA ha emergido como una herramienta de gran
potencial para abordar preguntas ecológicas a gran escala y alta resolución (Galaz García et al. 2023;
Veiga Branco et al. 2023).
La IA permite procesar grandes volúmenes de información (el “Big Data”),
lo que resulta especialmente útil en el contexto de la ecología, donde cada vez
es más común integrar múltiples fuentes como imágenes satelitales, sensores
remotos, o datos procedentes de estudios de campo (Buchelt
et al. 2024).
Pese al extenso uso de la
palabra “inteligencia artificial”, actualmente su definición sigue siendo algo
difusa. El concepto “IA” incluye todo sistema computacional construido para
realizar alguna tarea típicamente humana. Estas tareas pueden ser el
reconocimiento de objetos, toma de decisiones o el aprendizaje. Debido que la
mayoría de las aplicaciones implican procesos de aprendizaje, habitualmente se
usa el término aprendizaje automático (“machine learning”) como sinónimo
a la IA (Fig. 1). Sin embargo, técnicamente, el aprendizaje
automático únicamente engloba aquellos algoritmos de IA en los que su
rendimiento aumenta con la exposición a los datos. Dentro de la categoría de
aprendizaje automático existen diferentes grupos de algoritmos como el
aprendizaje profundo (“deep learning”), que a su vez incluiría la
subcategoría de la inteligencia artificial generativa que tanta popularidad ha
ganado en el último año (p. ej., Agathokleous et al. 2023; Biswas 2023; Rodas-Trejo y Ocampo-González 2024).
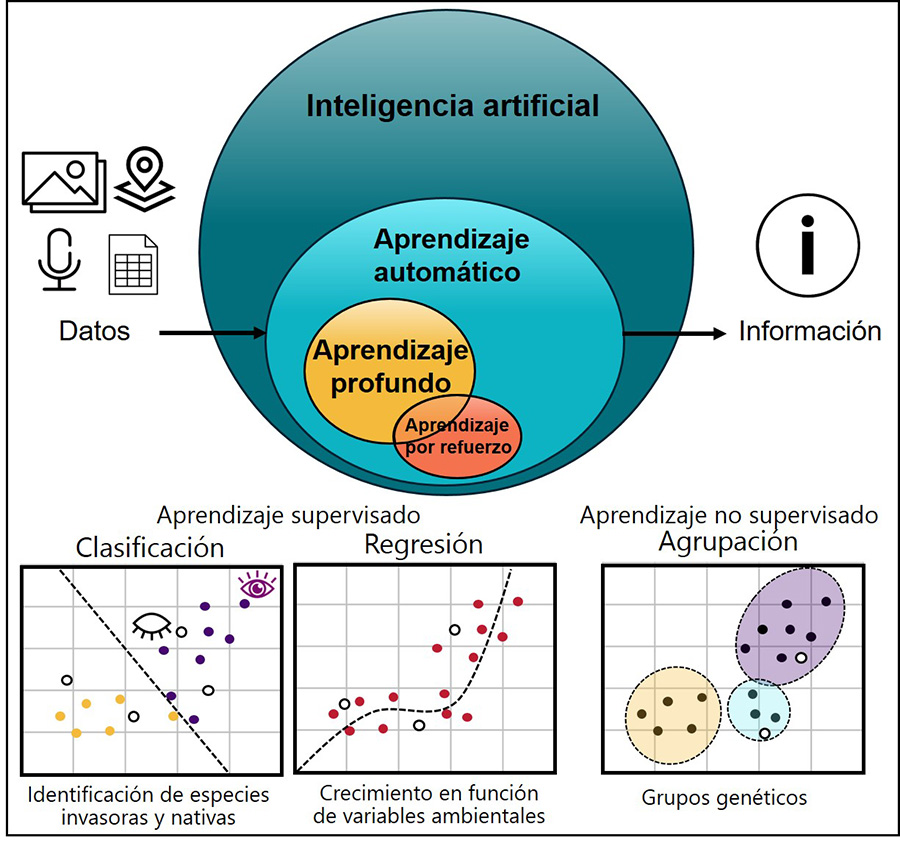
Figura 1. Inteligencia artificial (IA) para
principiantes. Diagrama de Venn mostrando las relaciones entre los términos:
Inteligencia artificial, aprendizaje automático, profundo y por refuerzo, así
como las tareas de aprendizaje supervisado para tareas de clasificación y
regresión (con datos etiquetados), y no supervisado (datos no etiquetados) para
tareas de agrupación. En los tres gráficos, los puntos blancos simbolizan los
datos de prueba, que el modelo (en línea discontinua) no ve durante el
entrenamiento del modelo y sirven para comprobar si el modelo está o no sobre
ajustado a nuestros datos o si es generalizable. Debajo de cada gráfico se
presenta un ejemplo de aplicación en invasiones biológicas.
Figure 1. Artificial intelligence (AI) for Beginners. Venn diagram
illustrating the hierarchy of the presented terms: Artificial Intelligence,
Machine Learning, Deep Learning, and Reinforcement Learning, as well as
supervised learning tasks for classification and regression (using labelled
data) and unsupervised learning (using unlabelled data) for clustering tasks.
In all three graphs, white dots represent test data. Dashed lines represent the
model. Below each plot, we provide an example of application in biological
invasions.
El funcionamiento de los algoritmos de aprendizaje
automático se basa en la exposición ordenada a los datos, de forma similar al
aprendizaje humano. Los datos se dividen generalmente en conjuntos de
entrenamiento, validación y prueba, para asegurar que el modelo aprendido por
la computadora generalice bien a nuevos datos (Greener
et al. 2022). Este proceso se conoce en la disciplina de aprendizaje
automático como adaptación de dominios (“domain adaptation”; Orouji et al. 2024). El conjunto de entrenamiento es el
grupo de datos con el que el modelo “aprende”, ajustando sus parámetros para
minimizar el error en las predicciones. Una vez que el modelo ha sido
entrenado, habitualmente se evalúa su rendimiento en el conjunto de validación,
un subconjunto de datos que permite ajustar y optimizar parámetros del modelo (Hastie et al. 2009). Finalmente, el conjunto de prueba
es un grupo de datos completamente nuevo para el modelo, utilizado solo al
final para medir su capacidad de generalización y obtener una evaluación
precisa de su rendimiento en datos no vistos. Hablamos de sobreajuste (“overfitting”)
cuando el rendimiento del modelo empeora al evaluarse con los datos de prueba (Greener et al. 2022).
La forma en que dividimos los datos en los diferentes
conjuntos balancea el rendimiento del modelo y su capacidad de generalización y
predicción (Walsh et al. 2021). Una proporción habitual
para dividir los datos es el “90-10” (Bzdok et al. 2018),
donde el 90 % de los datos se destina al entrenamiento del algoritmo de IA y el
10 % a su prueba de rendimiento/predicción. Estas proporciones pueden ajustarse
según el tamaño del conjunto de datos y debe considerar todo el rango de los
datos. Con conjuntos de datos pequeños, se recomienda utilizar técnicas como la
validación cruzada (Hastie et al. 2009) para
maximizar el uso de los datos disponibles y obtener una evaluación más precisa.
Otro método de validación común es la validación con bootstrapping (Tsamardinos et al. 2018), que selecciona múltiples
subconjuntos de datos de manera aleatoria y con reemplazo, evaluando el modelo
en cada subconjunto y promediando los resultados.
Para determinar si en una fotografía aparece una especie
invasora en particular, es necesario contar con muchas imágenes etiquetadas que
indiquen si la especie está presente o no. Publicaciones recientes utilizando
estas técnicas incorporan desde 300 (Mqingwana et al.
2024), 4200 (Matsuhashi et al. 2024) o hasta
309 373 fotografías (Chaity y van Aardt 2024).
Una vez que hemos examinado parte de esas fotografías (conjunto de
entrenamiento) y sus atributos relevantes (como la forma y disposición de las
hojas, el tamaño, el color de la flor, etc.), seremos capaces de identificar si
en una nueva foto (conjunto de prueba) esa especie está o no presente. Cuando
esta tarea la realiza un sistema computacional, hablamos de aprendizaje
supervisado, ya que sabiendo la categoría de cada imagen (presencia o no de la
especie), supervisamos cómo realiza la clasificación el sistema de IA. En
cambio, si no tenemos información sobre qué especies aparecen en cada
fotografía (datos no etiquetados), el modelo no podrá aprender a identificar la
presencia de una especie específica, pero sí podrá agrupar las imágenes en
función de similitudes, como el tipo de planta (herbácea, arbusto, árbol) o el
color de las flores. Este tipo de aproximación es un caso de aprendizaje no
supervisado, ya que el algoritmo detecta patrones de agrupación basándose
únicamente en datos no etiquetados.
Dentro del aprendizaje automático, hay diversas tareas que
permiten analizar y extraer patrones de los datos según los objetivos del
estudio. Para modelos de aprendizaje supervisado, una de las más comunes es la clasificación
(Greener et al. 2022; Walsh et al. 2021), donde el modelo aprende a asignar
categorías o etiquetas a nuevos datos. Por ejemplo, categorizar la especie de
ave en función de su canto (Knight et al. 2017) o
detectar la presencia de carrizo invasor (Phragmites australis; Xiong et al. 2024). Otra tarea es la regresión (Greener et al. 2022), utilizada para predecir valores
continuos, como la probabilidad de invasión en función de variables ambientales
(Pasha y Reddy 2024), o la cantidad de especies
exóticas en un ecosistema (Toussaint et al. 2014).
Los algoritmos más utilizados para estas tareas son la regresión logística o
lineal, máquinas de soporte vectorial (“support vector machines” [SVM]; Ben-Hur et al. 2008), árboles de decisión (incluyendo el
más famoso: Random forest; Simon et al. 2023) y
K-vecinos más cercanos (“K-Nearest Neighbors” [KNN]; Bzdok
et al. 2018).
Para modelos de aprendizaje no supervisado, las tareas más
comunes son la agrupación y la asociación. La agrupación (“clustering”; Jain et al. 2010) es una técnica que permite agrupar datos
en función de su similitud sin necesidad de etiquetas previas. La tarea de asociación
o afinidad busca descubrir relaciones entre variables o características dentro
de un conjunto de datos (Anandhavalli et al. 2010).
Tanto algoritmos de agrupación como afinidad han sido utilizados para
identificar asociaciones de fitoplancton, definiendo grupos funcionales (Zhu et al. 2022), para identificar condiciones aceptables
en el contexto de la restauración ecológica (Hamilton
y Murphy 2020) o para delimitar especies a nivel taxonómico (Pyron 2023). Los algoritmos utilizados para estas tareas se
dividen en métodos de aglomeración jerárquica y métodos que requieren la
definición del número de grupos, como k-means (Greener
et al. 2022).
Además de estos dos tipos de aprendizaje (supervisado y no
supervisado), existen opciones mixtas, como el aprendizaje semi-supervisado y
el aprendizaje por refuerzo. En el aprendizaje semi-supervisado (Hastie et al. 2009) se emplea una combinación de datos
etiquetados y no etiquetados, como por ejemplo para reconocer biodiversidad
acuática con pocos registros etiquetados (Ma et al. 2024).
En cambio, el aprendizaje por refuerzo (“reinforcement learning”; Lapeyrolerie et al. 2022) introduce un elemento
adicional: la recompensa. En este tipo de aprendizaje, el modelo toma acciones,
recibe información del entorno resultante y una evaluación de sus decisiones. Ha
sido especialmente de interés para la toma de decisiones de conservación (Lapeyrolerie et al. 2022), como la optimización de
recursos en gestión de fauna (Fonnesbeck 2008) o
balancear costes y beneficios de la protección de la biodiversidad en
diferentes áreas (Silvestro et al. 2022).
Existen diferentes aproximaciones para el aprendizaje en
función del nivel de transparencia del modelo. Las tareas de regresión,
clasificación, agrupación y asociación se pueden resolver con diferentes
aproximaciones en función de si especificamos las variables a partir de las que
queremos que el modelo aprenda (aprendizaje automático), o si dejamos que el
modelo identifique las variables más relevantes (aprendizaje profundo). En el
aprendizaje profundo (“deep learning”; Christin
et al. 2019; Borowiec et al. 2022), el
algoritmo detecta y extrae los atributos más relevantes de nuestros datos
gracias a la descomposición de estos en múltiples capas y nodos que se
relacionan de una forma similar a una red neuronal (Olden
et al. 2008). Las redes neuronales convolucionales (“convolutional
neural network” [CNN]) y las redes neuronales recurrentes (“recurrent
neural networks” [RNN]) son las arquitecturas de redes neuronales más
usadas en ecología (Christin et al. 2019) y pueden
ser adaptadas tanto a tareas de regresión como clasificación. Algunos ejemplos
en ecología incluyen la cuantificación del daño foliar (Loyani
2024) o la detección de peces a partir de videos submarinos (Fleure et al. 2024). Especialmente, en aprendizaje
profundo, se ha popularizado el uso de aprendizaje por transferencia (“transfer
learning”; Pan y Yang 2010), en el que se
reutiliza o adapta unos datos nuevos a un modelo previamente entrenado con
otros datos. Por ejemplo, bases de datos de imágenes públicas como las
disponibles en ImageNet (Deng et al. 2009) pueden ser
utilizadas para pre-entrenar un modelo descargable en línea que después se
puede utilizar para identificación de tipos de plancton a través del paquete de
R EcoTransLearn (Wacquet y Lefebvre 2022).
Existen diferentes opciones para
elegir el mejor algoritmo una vez identificada la tarea a realizar. Una de las
aproximaciones más utilizadas es comparar baterías de algoritmos, seleccionando
el que resulta en mayor precisión (Cao et al. 2020).
Otra aproximación es el ensamblaje de múltiples algoritmos para un resultado
más robusto, combinando las ventajas de cada modelo individual. Los métodos de
en ensamblaje más comunes son el bagging (promedio de múltiples modelos
entrenados en subconjuntos de los datos) y el boosting (entrenamiento
secuencial de modelos) (Dietterich 2000).
Los algoritmos de aprendizaje automático pueden tomar una
gran variedad de tipos de datos. Por lo general, al disponer de variables
específicas para el entrenamiento (en contraposición a archivos complejos como
imágenes, videos o audios) el modelo resulta más interpretable, permitiendo
examinar los atributos que son más relevantes para la predicción. Sin embargo,
esto requiere un alto conocimiento del problema y un gran procesamiento de
datos. Por ejemplo, para detectar diferentes clases de plancton a partir de
imágenes, Orenstein et al. (2022) obtuvieron el
tamaño, la forma y arquitectura, la presencia de extensiones (espinas u otras
estructuras defensoras), transparencia, color o presencia de reservas
lipídicas, a partir del examen individual de cada fotografía, y sugirieron el
uso de aprendizaje profundo para reducir el tiempo de procesado manteniendo la
precisión del modelo. La realidad detrás del uso de aprendizaje automático es
que la gran mayoría del tiempo se invierte en el procesado de los datos (“data
wrangling”) y no en la propia programación del modelo. Por ese motivo es
especialmente importante entender bien nuestros datos, plantear el objetivo de
nuestro modelo y escoger en consecuencia el modelo a utilizar.
Estado del arte del uso de la IA en el estudio de
invasiones biológicas
El uso de la IA en el estudio de invasiones biológicas
avanza lentamente en comparación con su aplicación en otras áreas de la
ecología. Para comprender las tendencias en este campo, realizamos una revisión
sistemática de artículos publicados en revistas incluidas en la base de datos Science
Citation Index (SCI) que utilizan IA en el estudio de especies invasoras.
Empleamos los criterios de búsqueda de Han et al. (2023)
que tenía por objetivo capturar los artículos en ecología que incluían el uso
de la IA a través de la búsqueda de las palabras clave “artificial
intelligence” o “machine learning” y en las áreas de “ecology”
o “environmental sciences”. No obstante, ampliamos los términos para
incluir una mayor diversidad de algoritmos y palabras clave relacionadas con
especies invasoras. La búsqueda realizada en Web of Science
(WoS) fue: “TS = (("invasive species" OR invader* OR "alien
species" OR "non-native" OR neophyte* OR neobiota* OR
"invasive alien species" OR exotic* OR invasion OR "biological
invasion" OR "invasive organisms") AND ("machine
learning" OR "artificial intelligence" OR "deep
learning" OR "neural network*" OR "support vector
machine" OR "random forest" OR "reinforcement
learning" OR "genetic algorithm" OR "supervised
learning" OR "unsupervised learning")) AND PY = (1997-2024) AND
DT = (Article) AND SU = (Agriculture OR Environmental Sciences OR
Multidisciplinary Sciences OR Biochemistry Molecular Biology OR Geosciences
Multidisciplinary) NOT TS = (cancer OR human OR medic* OR optical OR photon OR
metasurface OR protein*)”. La búsqueda, realizada el 30 de octubre de
2024 a través de Web of Science (WoS), identificó 415 artículos, que
fueron posteriormente filtrados para excluir duplicados y estudios fuera del
área de interés de este manuscrito. Algunos estudios
fuera del área de interés fueron publicaciones relacionadas con procesos de
invasión celular o el desarrollo de nuevas metodologías no invasivas. Tras
esta criba, retuvimos 278 artículos. Para identificar el crecimiento del uso de
la IA en las invasiones biológicas, detectar patrones en los modelos, tareas o
grupos taxonómicos en los que se ha utilizado, de estos 278 artículos
seleccionados, extrajimos información sobre especies estudiadas, algoritmos
aplicados y objetivos del uso de la IA. Entre las diferentes aplicaciones de IA
agrupamos los artículos en: detección, modelaje de la invasión (determinantes
de la presencia de la especie, identificación de rasgos relacionados con la
capacidad invasora, cuantificación de impactos), predicción de hábitat, y otras
aplicaciones (generar datos de prueba, desarrollo de aplicaciones comerciales,
análisis de literatura y simulaciones de gestión) (Fig. 2).
La producción de artículos sobre IA e invasiones ha crecido
significativamente en los últimos años. Aunque el uso de la IA en este campo
data de 1999, casi la mitad de los artículos (48.16 %) se han publicado entre
2022 y 2024 (Fig. 2A). Dichos estudios se han
centrado principalmente en tres grupos taxonómicos: plantas (45.3 %),
artrópodos (20.9 %) y peces (10.8 %) (Fig. 2B).
Este sesgo taxonómico coincide con las tendencias globales de especies
invasoras, donde las plantas son el grupo más numeroso, seguidas por insectos y
otros taxones, como crustáceos y moluscos (Roy et al. 2024).
En la última década, la detección de especies invasoras ha
sido la aplicación más destacada de la IA. Este crecimiento se refleja en el
aumento exponencial de publicaciones, con un máximo de 45 artículos en 2024, a
pesar de ser un año aún incompleto al momento de la revisión (30 de octubre; Fig. 2A). Las primeras aplicaciones de IA en
invasiones biológicas se centraron en el modelado y la predicción. Por ejemplo,
en 1999, Aussem y Hill utilizaron redes
neuronales para simular la expansión y biomasa del alga invasora comúnmente
llamada alga asesina (Caulerpa taxifolia) en el Mediterráneo. También en
esos años, Iguchi et al. (2004) construyeron modelos
de distribución para determinar el potencial invasor de diferentes especies de
perca utilizando un algoritmo genético entrenado con los datos climáticos de
las zonas que actualmente ocupan esas especies. Considerando todo el periodo,
1999-2024, las aplicaciones de la IA en invasiones biológicas han variado según
el taxón, con predominancia por la detección en plantas (p. ej., Marzialetti et al. 2021), modelado en peces (p.
ej., Epstein et al. 2018) y predicción en artrópodos
(p. ej., Gil-Tapetado et al. 2024) (Fig. 2C).
Los algoritmos más utilizados en los estudios revisados
incluyen modelos de regresión y clasificación. Los modelos de regresión han
sido aplicados para predecir la presencia de especies en función de variables
climáticas (p. ej., Yang et al. 2024 o Urza et al. 2024), mientras que los modelos de
clasificación han sido utilizados, por ejemplo, para identificar huevos de
peces invasores a partir de datos morfológicos (Camacho et al. 2019). En cambio, los modelos de
agregación y el aprendizaje por refuerzo han sido menos comunes. Entre los
modelos de agregación se encuentran aplicaciones como agrupar los patrones de
comportamiento de un caracol invasor en función de la temperatura (Bae et al. 2021). Solo cuatro estudios aplicaron
aprendizaje por refuerzo en nuestra búsqueda, principalmente en simulaciones
complejas, como interacciones entre especies (Prober et
al. 2012; Blonder et al. 2024) o modelos de
gestión óptima en ecosistemas acuáticos (Potapov 2009).
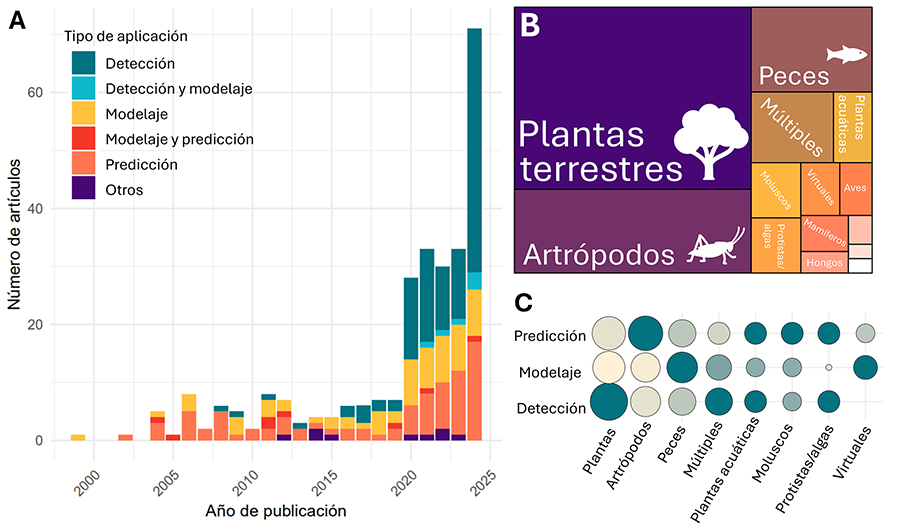
Figura 2. Resumen gráfico de la revisión
sistemática del uso de inteligencia artificial (IA) para el estudio de especies
invasoras en la literatura científica. A) Cronograma de los artículos
encontrados (1999-2024, búsqueda el 30 de octubre de 2024) y la clasificación
de la aplicación por la cual se utiliza la IA. B) Mapa de árbol
representando el número de estudios en diferentes grupos taxonómicos,
destacando los que agruparon mayor número de artículos: plantas, artrópodos y
peces. C) Mapa de puntos relacionando las diferentes tareas de la IA con
los taxones para los que se encontraron más de cinco artículos. Mientras que el
tamaño del punto representa el numero relativo de artículos totales, la
intensidad de color representa la tarea mayoritaria en cada grupo taxonómico
(que coincide con el tamaño de punto más grande para cada taxón).
Figure 2.
Graphical summary of the systematic review of the use of artificial
intelligence (AI) for studying invasive species in scientific literature. A) Published articles timeline
(1999–2024, search conducted on October 30th, 2024) and the
classification of applications for which AI was used. B) Tree map illustrating the number of
studies across different taxonomic groups, highlighting those with the highest
number of articles: plants, arthropods, and fishes. C) Scatterplot linking different AI tasks to the taxonomic groups
with more than five articles. The size of the points represents the relative
number of total articles, while colour intensity indicates the predominant task
for each taxonomic group (matching the largest point size for each taxon).
Descubrir: Métodos de detección de especies invasoras
con IA
La detección de especies invasoras es una de las
aplicaciones más importantes de la IA en la disciplina de las invasiones
biológicas, especialmente en los últimos años (Fig. 2).
La detección, y especialmente la detección temprana, es crucial en la gestión
de especies invasoras para reducir sus impactos a largo plazo (Latombe et al. 2017). Las mejoras en la captura (Nazir y Kaleem 2021) y procesado de imágenes (Wang et al. 2010; Krizhevsky et
al. 2017), así como la disponibilidad de imágenes digitales (Depauw et al. 2022) y satelitales (Cavender-Bares et al. 2022), han facilitado mucho
el desarrollo de múltiples herramientas para la identificación automática de
especies invasoras (Martinez et al. 2020).
La mayoría de los estudios de
detección que resultaron de la revisión sistemática realizada, se basaron en
imágenes como fuente principal de datos. Más del 90 % de los artículos revisados (correspondiente a 102 artículos) utilizaron
imágenes obtenidas de satélites (32.4 %), cámaras digitales (29.4 %), drones o
vehículos controlados remotamente (27.5 %) y tipos de cámaras o sensores
especializados (10.8 %; imágenes termales, cámaras hiperespectrales, LiDAR,
etc.). En el 9 % de artículos restantes se han empleado
datos alternativos como grabaciones de audio (Gugele
et al. 2021; Wood et al. 2024), trayectorias de
movimiento (Wiegleb et al. 2022) o características
morfológicas (Camacho et al. 2019).
Los algoritmos de aprendizaje profundo, especialmente las
CNN, son predominantes en la detección de especies invasoras. Estos algoritmos
destacan por su capacidad para procesar datos visuales sin necesidad de
variables predefinidas (García-Navarrete et al. 2024). Las CNN,
diseñadas específicamente para analizar imágenes, han sido utilizadas en
numerosos estudios para identificar plantas, insectos y animales invasores (p.
ej., Kulkarni y Minin 2023; Cardoso et al. 2024; Keerthinathan
et al. 2024). Es de especial interés el que las CNN se pueden construir con
diferentes arquitecturas (diferente número y estructura de capas y neuronas) y
existen algunas arquitecturas predefinidas de interés, como AlexNet (Zhuang et al. 2023), ResNet (Bao et
al. 2024), EfficientNet (Devi et al. 2023), o YOLO
(p. ej., Matsuhashi et al. 2024; Salcedo et al. 2024), las cuales son discutidas en más
extensión a continuación.
Las imágenes satelitales ofrecen una herramienta poderosa
para la detección de plantas invasoras. Estudios como el de Castillo et al. (2008) marcaron el inicio de esta
línea de investigación, utilizando imágenes de MODIS 250 m RGB para detectar la
planta acuática Lemna obscura. Hasta el momento, únicamente encontramos
el uso de imágenes de satélite para la detección de plantas invasoras,
probablemente por la naturaleza sésil de estas, que permite en principio
obtener ocurrencias completas en ciertas áreas a partir de imágenes de satélite
multiespectrales (Wang et al. 2013; Meerdink et al. 2024). La ventaja del uso de imágenes
de satélite respecto a métodos más tradicionales como censos poblacionales de
campo (Salguero-Gómez y Gamelon 2021) o
estudios fenológicos mediante observación directa (Fenner
1998), radica en la cobertura espacial y temporal, que permite monitorear
grandes áreas y realizar análisis estacionales (Cavender-Bares
et al. 2022; Katal et al. 2022). No obstante, una
desventaja de las imágenes de satélite multiespectrales es su actual resolución.
Actualmente, la máxima resolución se encuentra entre 0.3 y 0.5 m (EOSDA
LandViewer), pero la eficiencia de la detección de especies dependerá, no solo
del tamaño de estas sino también del contraste en los hábitats que ocupa. Por
ejemplo, se ha demostrado que puede ser útil para la detección de ballenas o
elefantes en entornos homogéneos, pero no para ñus en biomas complejos (Hollings et al. 2018). En algunas especies, el uso de
imágenes en diferentes momentos permitió una detección más precisa, como al
principio de la temporada de crecimiento y durante el proceso de senescencia en
praderas (Raab et al. 2018), o antes y durante la
floración de la acacia azul (Acacia saligna; Marzialetti
et al. 2021).
El uso de cámaras digitales (incluyendo cámaras montadas en
vehículos remotos) agrupa la mayor cantidad de artículos que usan IA para la
detección de especies invasoras (29.4 % cámaras digitales, 27.5 % vehículos
remotos). Por ejemplo, Yang et al. (2022) construyó una base de datos con 3000
imágenes de semillas de 12 especies invasoras para entrenar una CNN para
detectarlas en el tránsito global. En este trabajo,
los autores utilizaron una de las arquitecturas de CNN comentada anteriormente
y que ha ganado más fama últimamente, el modelo YOLO (“You Only Look Once”,
traducido como: solo miras una vez) que también se ha utilizado para
detectar la avispa asiática (Vespa velutina; O'Shea-Wheller
et al. 2024), la chinche hedionda (Halyomorpha halys; Palazzetti et al. 2024), o ciervo de Timor (Rusa
timorensis; Sudholz et al. 2022). Algunos de los
modelos comentados en el anterior párrafo utilizan no solo una arquitectura de
red neuronal predefinida, sino también una red pre-entrenada (aprendizaje por
transferencia), reduciendo así la demanda de datos. Este es el caso de Gupta et al. (2020) para la detección de plántulas no
deseadas en base a fotografías de RGB, o Ulhaq et al.
(2021) para detectar cerdos, conejos y canguros en base a imágenes
térmicas.
La disponibilidad de imágenes digitales en línea ha
permitido aplicaciones de detección de especies invasoras sin recogida de datos
en campo. Por ejemplo, las imágenes de Google Street View permiten
detectar especies invasoras a lo largo de carreteras (Kotowska
et al. 2021). También imágenes en redes sociales (Flickr, Twitter,
Instagram, y Facebook) han permitido la detección temprana de la hierba de la
pampa (Cortaderia selloana; Cardoso et al. 2024)
y datos de redes sociales en general también son especialmente útiles para
entender la dimensión social de las especies invasoras (Novoa
et al. 2025).
El uso de drones para el monitoreo de especies invasoras ha
incrementado rápidamente en la última década (Dash et al.
2019; Singh et al. 2024). En su revisión, Singh et
al. destacaron que la mayoría de los estudios hasta la fecha se centran en
probar diferentes algoritmos de aprendizaje automático y profundo para
clasificar especies invasoras, y menos estudios que presentan monitoreo de
estas. En este contexto, las imágenes de dron resultan especialmente útiles
para cubrir grandes extensiones en áreas remotas o poco accesibles (Fig. 3), obteniendo así de forma precisa la
cobertura vegetal (Kattenborn
et al. 2020; Bravo-Diaz et al. 2024). El
estudio de James y Bradshaw (2020) presenta una
metodología para la detección de plantas con drones durante el vuelo, donde en
base a redes neuronales y el desarrollo en la aplicación DJI SDK UXSDKDEMO (https://github.com/DJI-Mobile-SDK-Tutorials/Android-UXSDKDemo)
se muestra en pantalla y en vivo las áreas donde se encuentra la especie
objetivo, de alto interés para especies invasoras. No solo para detectar la
presencia de individuos sino también para su conteo, el uso de imágenes de dron
ha demostrado ser más eficiente que el conteo humano (Hodgson
et al. 2018).
Las imágenes térmicas o el uso de videos pueden proporcionar
más precisión que las imágenes digitales para detectar algunas especies
invasoras en particular. La detección de mamíferos u otros endotermos (Stone y Davis 2024) es especialmente eficiente a
través de imágenes térmicas (Ulhaq et al. 2021; Gonzalez et al. 2016). El uso de videos para la
detección de especies invasoras proporciona mucha resolución temporal, así como
un mayor número de imágenes totales (Gonzalez et al. 2016; Ulhaq et
al. 2021; Sudholz et al. 2022; Dyrmann et al. 2024). Los videos resultan especialmente
útiles para el monitoreo de organismos móviles, incluyendo animales marinos (Fleure et al. 2024) o mamíferos terrestres (Ulhaq et al. 2021) y también la detección en movimiento,
como la detección de plantas invasoras en carreteras a partir de videos de un
coche con alta velocidad (Dyrmann et al. 2024).
La detección de especies
invasoras es una de las aplicaciones más avanzadas de la IA en invasiones
biológicas, especialmente mediante el uso de imágenes. Satélites, drones y
cámaras digitales destacan como las principales fuentes de datos, mientras que
algoritmos de aprendizaje profundo, como redes neuronales pre-entrenadas,
lideran las metodologías. Las tecnologías emergentes, como imágenes térmicas,
videos y datos de redes sociales, han ampliado las capacidades de detección,
permitiendo identificar especies en territorios amplios, áreas remotas y
entornos dinámicos. Estos avances consolidan la IA como una herramienta
esencial para la gestión y el monitoreo de especies invasoras.

Figura 3. Detección, modelaje y predicción de
plantas invasoras a partir de imágenes de dron e inteligencia artificial (IA).
La captura de datos demográficos a partir de aplicar la IA en imágenes de dron
permite capturar el tamaño de la población manteniendo las distancias con
especies invasoras dañinas como la chumbera (A), acceder a terrenos
difíciles (B) y cubrir grandes extensiones (C). Gracias al
entrenamiento de modelos de aprendizaje automático podemos detectar nuevos
individuos, cuantificar tasas vitales (supervivencia, crecimiento y
reproducción), evaluar el éxito de programas de erradicación, identificar
patrones de distribución espacial o proyectar la dinámica poblacional en el
tiempo.
Figure 3.
Detection, modelling, and prediction of invasive plants using drone images and
artificial intelligence (AI). The use of AI on drone imagery enables the
capture of demographic data, allowing the estimation of population size while
maintaining safe distances from harmful invasive species such as prickly pear
cactus (A), accessing
difficult terrains (B),
and covering large areas (C). Through machine learning, it is possible to detect new
individuals, quantify vital rates (survival, growth, and reproduction), assess
the success of eradication programs, identify spatial distribution patterns, or
project population dynamics over time.
Modelar: importancia de variables y adquisición de datos
Prácticamente un 30 % de los artículos (de un total de 278) que
utilizan IA en nuestra revisión de la literatura lo hicieron para modelar algún
aspecto del proceso de invasión, sin incluir la proyección de su distribución. Estas
investigaciones abordan principalmente dos áreas: la contribución de conjuntos
de predictores para variables de interés y la mejora en la adquisición de
datos. Este enfoque incluye estudios que exploran atributos invasores,
demografía a partir de imágenes y análisis de impactos.
La IA permite identificar la importancia relativa de
predictores sobre variables de interés de manera eficiente. Los algoritmos de
aprendizaje automatizado facilitan el manejo de relaciones no lineales y la
identificación de factores clave en procesos biológicos. Entre las aplicaciones
destacadas en invasiones biológicas se encuentran la explicación de la presencia
o abundancia de especies (Sampaio Franco et al.
2021; White et al. 2022), la riqueza de especies
invasoras (Toussaint et al. 2014; Santoianni et al. 2024), la dispersión de especies (Pontin et al. 2011) y el éxito de programas de
erradicación (Xiao et al. 2018). También destacamos la
existencia de múltiples trabajos que se enfocan en identificar características
que determinan el comportamiento invasor mediante bases de datos de atributos (Kolar 2004; Keller et al. 2011; Philibert et al. 2011).
Aparte de determinar la importancia de factores, otra
aplicación de la IA para modelar las invasiones biológicas es ampliar o
acelerar la captura de datos. El aprendizaje automático y profundo se han
utilizado para entrenar modelos que permiten una adquisición de datos más
rápida y menos costosa con una alta precisión (Fig. 3).
El desarrollo de cámaras y lentes ha permitido plantear la substitución de las
largas visitas al campo por el vuelo de drones o los costosos análisis al
laboratorio por fotografías hiperespectrales. Por ejemplo, gracias al
desarrollo de métodos de reconstrucción 3D es posible estimar el volumen
corporal de animales de forma remota a través del vuelo de un dron (Los et al. 2023; Stone y Davis
2024). En invasiones biológicas se han empleado imágenes para caracterizar
el crecimiento y estructura poblacional (Benzer y
Benzer 2020; Bednarsek et al. 2022),
componentes esenciales para cuantificar las dinámicas de invasión. Otra fuente
de datos que la IA nos permite explotar mejor es la literatura publicada. Por
ejemplo, para entender las aproximaciones en la restauración forestal, Nunez-Mir et al. (2015) hacen una revisión sistemática
con análisis de contenido automático, lo que denominamos “big literature”
(Bolaños et al. 2024).
La IA se ha utilizado no solo para entender los
determinantes de la invasión, o cuantificar su demografía, sino también para
evaluar sus impactos. Por ejemplo, para cuantificar los impactos específicos
sobre otra especie, como cuantificar el nivel de daño foliar sobre hojas de
tomate por parte de la polilla del tomate (Tuta absoluta; Loyani 2024). De forma similar, el uso de la IA también
resulta útil para reducir datos multidimensionales, como la composición del
microbioma del suelo (Li et al. 2022) o propiedades
fisicoquímicas (Zhang et al. 2023) y encontrar
marcadores de interés que nos ayuden a detectar los impactos de la invasión.
La IA se utiliza en el modelado de procesos de invasión,
destacando su capacidad para identificar factores clave, acelerar la
adquisición de datos y evaluar impactos. Herramientas como drones, análisis
hiperespectrales y algoritmos avanzados han optimizado el estudio de atributos
invasores, demografía y evaluación de daños específicos. Además, la aplicación
de IA a grandes bases de datos, como literatura científica, amplía su potencial
para abordar las complejidades de las invasiones biológicas.
Predecir: Modelos de distribución de especies invasoras
Los modelos de distribución de especies (“species
distribution models” [SDMs]; Pearson 2003) son
herramientas fundamentales para predecir la idoneidad ambiental de las especies
(Guisan et al. 2017). Por esta razón, han sido
ampliamente empleados para predecir la distribución potencial de las especies
invasoras (Guisan et al. 2014). Los SDMs capturan la
relación entre los registros de presencia de especies y diferentes predictores
(p. ej., temperatura, precipitación o tipo de suelo), utilizando estas
relaciones para mapear la probabilidad de presencia de una especie invasora (Davis et al. 2024).
La mayoría de los estudios aquí revisados emplearon IA para
construir SDMs, aunque existen otras aplicaciones predictivas. De los 91
artículos sobre predicción, el 93 % utilizaron IA para SDMs, mientras que el
resto construyó predicciones basadas en cambios de uso del suelo (Martínez-Vega et al. 2017) o crecimiento bajo
diferentes condiciones ambientales (Javed et al. 2022).
El interés de los SDMs reside en el gran potencial que tienen incluso cuando
disponemos de pocos datos, permitiendo construir el nicho ecológico de especies
invasoras y cuantificar cambios en este, o el potencial invasor en nuevas áreas
(Jeschke y Strayer et al. 2008; Barbosa et al. 2012).
El uso de algoritmos para SDMs ha evolucionado, reflejando
cambios en las tecnologías disponibles, desde los algoritmos genéticos al uso
de baterías de algoritmos y su ensamblaje. En 2008, Jeschke y Strayer revisaron
los métodos estadísticos más utilizados para SDMs, destacando, entre otros, la
regresión logística, los modelos lineares generalizados (Lehman
et al. 2002), los modelos aditivos generalizados (Leathwick
et al. 2006), los árboles de regresión y clasificación (Elith
et al. 2006), las redes neuronales (Pearson et al.
2002) y los algoritmos genéticos (Stockwell
y Peters 1999). Sin embargo, en 2012, Barbosa et al. destacaban que el
algoritmo genético GARP (“Genetic Algorithm for Rule-set Production”; Stockwell y Peters 1999) fue el más usado para
el mismo propósito. Este algoritmo se basa en los principios de la selección
natural para determinar un conjunto de atributos que mejor optimizan la
variable a predecir, incluyendo procesos de cruce de atributos y mutación (Stockwell y Peters 1999). Debido a su uso
extendido, existe un paquete de R especifico (GARPTools) para la
construcción y evaluación de SDMs con GARP (Haase et al.
2021).
En nuestra revisión sistemática encontramos la predominancia
del uso de un solo algoritmo de aprendizaje automático, especialmente GARP y
también Maxent (“Maximum Entropy Modelling”; Phillips
et al. 2006). Maxent es un método de modelado estadístico que busca la
distribución más uniforme (máxima entropía) que se ajuste a los datos de
ocurrencia y a las restricciones ambientales disponibles. Maxent es popular en
la comunidad ecológica por su capacidad para trabajar con datos de presencia
únicamente y por generar modelos robustos incluso con conjuntos de datos
incompletos o limitados (Jiang et al. 2018; Adepoju et al. 2020; Morey y Venette 2020). A partir de aproximadamente
2016 ya no hay artículos usando GARP, mientras que algoritmos basados en
arboles de decisiones como los random forest y redes neuronales ganan
terreno, así como el uso de baterías de algoritmos en vez de uno de solo. Por
ejemplo, Pasha y Reddy (2024) ajustaron múltiples
modelos incluyendo Maxent, modelos lineales generalizados, modelos aditivos
generalizados, redes neuronales, modelo potenciado generalizado y random
forest, seleccionando el que presenta mejor ajuste para predecir la
distribución del arbusto mezquite (Prosopis juliflora) a distintos
escenarios de cambio climático. Otros estudios no solo compararon todos estos
modelos, sino también ensamblan sus resultados para producir una distribución
final. Tal es el caso de Mathur y Mathur (2024),
que utilizaron ensamblaje de modelos para predecir la distribución de la
chumbera (Opuntia ficus-indica) bajo múltiples escenarios de cambio
climático. Los autores incluyeron modelos lineales generalizados, modelos
aditivos generalizados, support vector machine, arboles de decisión, random
forest, Maxent, MARS (“multivariate adaptive regression splines”) y
redes neuronales, combinando las proyecciones resultantes de cada modelo en una
sola capa promedio. Es también el caso de Nisin et al.
(2023) que proyectó la distribución del pez piracú (Arapaima gigas)
a través de promediar las probabilidades de presencia de la especie de una
selección de los mejores modelos entre 10 algoritmos.
Aunque útiles, los SDMs presentan limitaciones importantes
que deben considerarse (Elith et al. 2011). Existen
múltiples artículos revisando cómo construir SDMs de forma apropiada (p. ej., Araújo et al. 2019; Zurell et al.
2020; Sillero et al. 2021; Hui
2023; Soley-Guardia et al. 2024; Davis et al. 2024). Sin embargo, estos modelos se basan
en correlaciones, no en causalidad, y son sensibles a los datos de ocurrencias,
que suelen ser imperfectos (Chen
et al. 2013). Por ello, los SDMs mecanicistas, que incorporan procesos
ecológicos explícitos, son una alternativa prometedora, especialmente para
especies invasoras (Fenollosa et al. 2025), aunque
requieren una alta disponibilidad de datos (Briscoe et
al. 2019).
Gestión automatizada de especies invasoras con IA
La inteligencia artificial proporciona herramientas clave
para la gestión de especies invasoras, abordando desafíos complejos con mayor
eficacia y precisión. Las capacidades analíticas y predictivas previamente
discutidas encuentran aplicaciones prácticas en la detección temprana, la
delimitación de áreas afectadas, y el diseño de estrategias de control y
erradicación. Estas herramientas están transformando la gestión de invasiones
biológicas al optimizar la priorización de acciones y reducir costos operativos.
A continuación, examinamos cómo estas tecnologías están revolucionando las
prácticas de manejo y control de especies invasoras.
La detección temprana es una de las aplicaciones más
efectivas de la IA en la gestión de especies invasoras. Herramientas como el
monitoreo acústico en tiempo real se han implementado para rastrear la
expansión del búho barrado (Strix varia) en América del Norte (Wood et al. 2024). Además, los modelos de distribución han
ayudado a identificar áreas de riesgo, como espacios protegidos (Dar et al. 2023) o regiones afectadas bajo escenarios de
cambio climático (Zhao et al. 2023a), priorizando así
los esfuerzos de gestión.
El procesamiento avanzado de imágenes ha permitido el
desarrollo de herramientas autónomas para la identificación y control de plantas
invasoras. En el ámbito agrícola, productos comerciales emplean imágenes de
alta resolución (AutoWeed, https://autoweed.com.au/),
hiperespectrales (Ladybird, Universidad de Sidney, https://robohub.org/robots-ladybird/)
o termales (Weedseeker 2, Trimble Agriculture, https://ptxtrimble.com/es)
para la detección y eliminación precisa de hierbas no deseadas (Roberts y Florentine 2024). En ecosistemas
acuáticos, un pequeño barco no tripulado utiliza aprendizaje profundo basado en
YOLO para detectar y recolectar automáticamente algas o plantas acuáticas invasoras,
como Lemna minor, a través de videos capturados en tiempo real (Salcedo et al. 2024). De
forma similar, Patel et al. (2019) usan redes
neuronales para detectar múltiples plantas acuáticas invasoras a partir de
señales hidroacústicas tomadas bajo el agua, y lo acoplan a pequeños barcos
autónomos que liberan pequeñas dosis de herbicida al encontrar las plantas
objetivo.
La IA también se emplea en soluciones interactivas y
específicas para gestionar fauna invasora. Un ejemplo es el uso de sistemas de
captura con cebo automatizado, como el desarrollado por Charlton
et al. (2024), que utiliza algoritmos para clasificar especies invasoras y
dispensar cebos personalizados para fauna invasora. Asimismo, tecnologías
basadas en aprendizaje profundo permiten ahuyentar aves invasoras mediante la
detección de su canto y la reproducción de sonidos específicos para proteger
cultivos (Chen et al. 2024). El uso de robots basados
en animales permite la interacción directa y promete ser clave para la
conservación de la biodiversidad (Schmickl y
Romano 2024). Por ejemplo, se han utilizado robots mimetizando predadores
para proteger renacuajos nativos de peces invasores como la gambusia (Gambusia
affinis) (Polverino et al. 2022).
Las tecnologías basadas en IA facilitan la detección de
introducciones o reintroducciones a través de aplicaciones innovadoras. Por
ejemplo, Yang et al. (2022) entrenó un modelo para
identificar semillas invasoras mediante imágenes captadas en controles
fronterizos, mientras que Kulkarni y Di Minin
(2023) han utilizado IA para rastrear el comercio ilegal de fauna a partir
de fotografías en plataformas digitales.
La IA transforma la gestión de especies invasoras mediante
herramientas que permiten la detección temprana, la interacción en tiempo real
y la automatización de acciones específicas. Tal y como hemos presentado en
esta sección, robots y sistemas autónomos con IA facilitan la gestión a tiempo
real, mientras que el uso de aprendizaje profundo y análisis de datos mejora la
vigilancia continua y la evaluación de los esfuerzos de erradicación. Estas
innovaciones no solo aumentan la eficiencia de las intervenciones, sino que
también reducen significativamente los costos operativos, posicionando a la IA
como un componente esencial en la lucha contra las invasiones biológicas.
Paso a paso usando IA en tu proyecto con especies
invasoras y practicas éticas
El ciclo de vida de los proyectos de datos proporciona un
marco estructurado para implementar la IA (Fig. 4).
Este proceso circular, regresivo en ocasiones, comienza con la delimitación del
problema y finaliza con la comunicación de resultados. En este apartado, se
examinan los componentes clave de este ciclo, destacando consideraciones
específicas como fuentes de datos y aspectos éticos relevantes para las
invasiones biológicas.
La delimitación del problema es el primer paso y determina
el enfoque de todo el proyecto de IA. Es crucial definir la tarea a resolver
(clasificación, regresión o agregación), el tipo de datos necesarios y los
algoritmos a utilizar. Es importante describir qué categorías vamos a querer
clasificar si nuestra tarea es de clasificación y el rango de la variable de
interés si nuestra tarea es de regresión, en relación con nuestros datos. Por
ejemplo, clasificar colores de flores de una especie invasora requeriría
categorías específicas de los colores posibles y dependiendo del tipo de datos
utilizados, una categoría adicional de “sin flor”, o hasta podríamos usar el
ángulo de color o matiz (en inglés: Hue) como definición numérica del color y
explorar qué factores contribuyen a la escala de color a través de la
regresión. Los análisis exploratorios pueden llevar a ajustes en el modelo,
subrayando la importancia de mantener flexibilidad en el diseño experimental.
Una vez planteado el problema y el diseño experimental
procederemos a la colección de datos. Como hemos visto en la revisión
sistemática, muchos estudios hacen uso de bases de datos o imágenes de acceso
libre, permitiendo extender las dimensiones temporales y espaciales de los
proyectos (Coughlan et al. 2020; Heger et al. 2021) (Tabla 1).
Estas bases de datos proporcionan datos de la ecología, estadio invasor, fechas
de introducción, atributos o impactos descritos de especies invasoras, que
podemos utilizar para análisis comparativos (Tabla 1).
Otras fuentes de datos que son de interés para el monitoreo de especies
invasoras son las imágenes satelitales e imágenes de redes sociales. En España
destacamos el centro nacional de información geográfica (www.centrodedescargas.cnig.es) que centraliza bases de
datos de imágenes satelitales. Adicionalmente, hace poco se desarrolló una base
de datos de imágenes de dron, GeoNadir (https://geonadir.com/)
que podrían proporcionar mayor resolución en sitios específicos, especialmente
para el entrenamiento de detección vegetal. Debido a la importancia de la
especie humana en las invasiones biológicas, datos de redes sociales,
tendencias en Google o comercio electrónico pueden ser especialmente útiles
para el monitoreo de procesos de invasión (Novoa et al.
2025). Para la captura de datos en portales web recomendamos las técnicas
de raspado web (Web scrapping) que permite la extracción de contenido
HTML de múltiples páginas de forma automática como a partir de anuncios de
venta de plantas para detectar tráfico ilegal (Maher et
al. 2023).
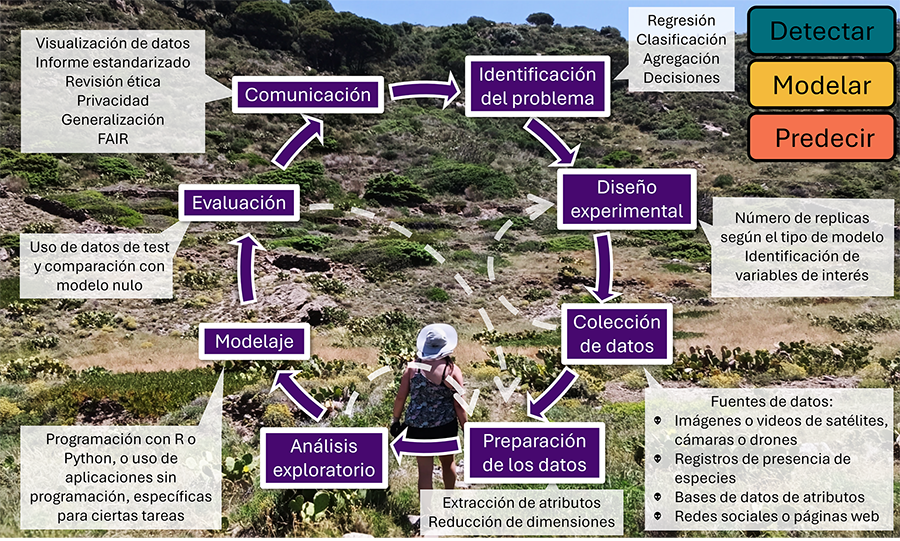
Figura 4. Elementos clave en el ciclo de vida
en los proyectos de datos para el estudio de especies invasoras, desde la
identificación del problema y la aplicación de la inteligencia artificial (IA)
(detectar, modelar, predecir), hasta la comunicación de resultados, que puede
llevar a la identificación de nuevas preguntas. El diagrama muestra los
elementos más importantes para la integración de la IA en nuestra
investigación. Para generalidades sobre un diseño experimental adecuado en
ecología recomendamos Marshall (2024).
Figure 4. Key elements in data science lifecycle for the study of invasive
species. The lifecycle spans from problem identification and artificial
intelligence (AI) application (detection, modelling, prediction) to the
communication of results, which may lead to the formulation of new questions.
The diagram shows the most important elements for the integration of AI in our
experiment. For generalities on appropriate experimental design in ecology we
recommend Marshall (2024).
La elección del enfoque
de aprendizaje condiciona los resultados y la metodología del proyecto. Mientras el aprendizaje profundo es ideal para
datos complejos como imágenes, requiere grandes volúmenes de datos. En cambio,
el aprendizaje automatizado es más transparente y adecuado para entender
condicionantes, pero podría necesitar un procesamiento previo de las variables
de interés. Para el análisis de imágenes existen algunos complementos para
automatizar esta extracción de atributos para entrenar modelos de aprendizaje
automático como el CIMMYT MaizeScanner en ImageJ (https://github.com/sckefauver/CIMMYT)
o los paquetes patternizes (Van Belleghem et al. 2018), pliman (Olivoto 2022) o recolorize (Weller et al. 2024) en R. En problemas más simples, o con pocos
datos, los métodos estadísticos tradicionales pueden ser más eficaces (Ley et al. 2022).
Diferentes lenguajes de programación destacan en su
potencial para ajustar modelos de IA. Programas como R, Python o MATLAB son los
más utilizados debido a la gran comunidad de usuarios y al desarrollo de
paquetes o librerías específicos para la implementación de algoritmos de
aprendizaje automático y profundo. Entre los programas de acceso libre, R solía
recoger la mayor parte de la comunidad de ecólogos por lo que encontraremos
mucho material de guía especializado, pero Python dispone de librerías mucho más
adecuadas para el entrenamiento de modelos con imágenes. Para ambos programas
hay una gran cantidad de ejemplos de aplicaciones de IA sobre diferentes tipos
de datos en el portal de competiciones de Kaggle, incluyendo ejemplos con
especies invasoras (https://www.kaggle.com/competitions/invasive-species-monitoring/).
Si no disponemos de un ordenador capaz de procesar cierta cantidad de datos,
recomendamos el uso del portal de Google Colab, que nos permite codificar en
diferentes lenguajes de programación y procesar los datos en línea, sin
limitación por la GPU de nuestro ordenador.
Las aplicaciones sin
programación amplían el acceso a la IA, haciendo más inclusivo su uso en
invasiones biológicas. En caso de que no tengamos experiencia en programación
existen múltiples aplicaciones específicas con interfaz gráfica que nos
permiten el uso de aprendizaje automático y profundo sin necesidad de
programar. Empezando por las aplicaciones más específicas, encontramos que
algunos artículos de la revisión sistemática realizada incluyeron una
aplicación web que permite el uso directo del modelo ya entrenado, como WhoseEgg
que permite subir datos de morfología de diferentes huevos y en base a un
modelo ya entrenado clasifica la especie y si es invasora o no (Goode et al. 2023). De forma similar, Palazzetti et al. (2024)
presenta una aplicación para Android que permite la detección a tiempo real de
la chinche hedionda (H. halys) a partir de imágenes usando un modelo
entrenado con redes neuronales. De forma más general, no específico para
plantas invasoras, destacamos que existen complementos que podemos añadir a
herramientas de análisis espacial para segmentar individuos invasores a partir
de imágenes, como por ejemplo el complemento de acceso abierto para QGIS GeoSAM
basado en el famoso modelo “segment anything” (Zhao
et al. 2023b; Prado Osco et al. 2023). Un
último ejemplo del potencial de aplicaciones concretas para acortar distancias
con la IA son portales como liner.ai o Roboflow. Estos portales
con modelo freemium (parte de los servicios son gratuitos) permiten de
forma muy simple entrenar diferentes algoritmos en base a imágenes, con una
interfaz muy amigable y permitiendo el etiquetado colaborativo de imágenes.
Tabla 1. Selección de
bases de datos de interés para proyectos de inteligencia artificial (IA) con
especies invasoras, incluyendo bases de datos específicamente para especies
exóticas e invasoras (en rojo), pero también bases de datos globales que
incluyen especies invasoras (turquesa). Abr.: abreviación.
Table 1. Selection of databases of interest to develop artificial
intelligence (AI) projects with invasive species, including databases that
target exotic and invasive species specifically (red) but also global databases
that include invasive species (turquoise). Abr: abbreviation.
El uso de la IA es cada vez una tarea más sencilla, pero no
está exenta de responsabilidad. Los patrones que observamos gracias a los
modelos de IA dependen (y se limitan) a las variables y datos que hemos
considerado y por ende están sujetos a sesgos. Este agosto de 2024 entró en
vigor el primer marco jurídico sobre IA en Europa (Ley de AI de la UE,
Reglamento 2024/1689) que ahora se está implementando en los estados miembros.
En este marco se ha redactado el Código de buenas prácticas de IA de propósito
general (GPAI) que se publicará durante la primavera de 2025. Sin embargo,
queremos destacar algunos aspectos esenciales, específicamente sobre el uso de
algoritmos de aprendizaje automático y considerando los tipos de datos
mayoritariamente usados en el campo de las invasiones biológicas.
Entre buenas prácticas podemos encontrar aspectos técnicos
de la construcción de los modelos, aspectos de representatividad de los datos,
aspectos de calidad e interpretación del modelo, consideraciones éticas y de
privacidad y principios FAIR (Fáciles de encontrar, accesibles, interoperables
y reutilizables) (Heil et al. 2021).
En cuanto a los aspectos técnicos, algunos aspectos
imprescindibles es asegurar que guardemos una parte de los datos representativa
para la comprobación a través de técnicas de muestreo estructurado y probando
diferentes particiones de datos. Otro aspecto técnico es la inclusión de muchas
variables colineales en algunos modelos de aprendizaje automático. Recomendamos
revisar las demandas de cada algoritmo específicamente, así como revisar los
parámetros del modelo analizando su impacto de forma estructurada (Fourcade 2021). Además, de forma general se recomienda
preparar el modelo de forma escalable, empezando con las partes más críticas y
añadiendo complejidad de forma progresiva.
Respecto la representatividad de los datos, es común que
estos no sean totalmente representativos de la realidad, sino que estén
sesgados por múltiples ejes. Por ejemplo, la huella colonialista sigue presente
en la actualidad, y es reconocible en los patrones de especies invasoras (Lenzner et al. 2022), impactando en la cantidad de
estudios y datos ecológicos disponibles y, por tanto, en la representatividad
de áreas con menos privilegios y realidades minoritarias (Trisos
et al. 2021). Recomendamos incluir análisis de representatividad para datos
sensibles (Clemmensen y Kjærsgaard 2022)
que delimitaran la posibilidad de generalización.
La interpretación del modelo es requisito indispensable en
proyectos con IA. Recomendamos contrastar las predicciones obtenidas con un
modelo nulo o previo y la revisión biológica de los resultados. Por ejemplo, el
uso SDM para la modelización de la presencia de especies de cuevas sin tener en
cuenta la disponibilidad de hábitat nos retornará predicciones especialmente
disparatadas (Mammola y
Leroy 2018). Durante la evaluación del modelo deberemos incluir diferentes
métricas en función del tipo de tarea realizada e informar no solo de la
precisión con los datos de prueba sino también en los datos de entrenamiento (Rainio et al. 2024).
La exposición de la herramienta de IA utilizada debe hacerse
de forma correcta, completa y clara, idealmente siguiendo los principios FAIR,
revisando políticas de privacidad y reconociendo el crédito de los datos usados
si no son de propia colección. Por ejemplo, es importante detallar la
responsabilidad del error de los modelos, por ejemplo, de
manejo automatizado (Dara et al. 2022) y evaluar si
nuestros datos incluyen datos sensibles como imágenes de personas no
informadas, por lo que su publicación requerirá el uso de protocolos de
encriptación (Jia et al. 2023).
Conclusiones y visión de futuro
La inteligencia artificial ha revolucionado el estudio de
especies invasoras, integrando innovación tecnológica con prácticas ecológicas.
Nuestra revisión sistemática muestra que la mayoría de los estudios enfocados
en IA y especies invasoras han priorizado tareas de detección, particularmente
en plantas. Esto se ha facilitado por la creciente disponibilidad de datos
abiertos, incluyendo imágenes satelitales, registros de redes sociales y datos
de drones, que permiten extender las dimensiones temporales y espaciales de los
proyectos. Desde la implementación de herramientas de detección temprana hasta
el uso de robots autónomos para el control de especies, la IA ha reducido
significativamente los costos y promete mejorar la eficiencia de los esfuerzos
de erradicación.
La disponibilidad y calidad de los datos son factores clave
que condicionan el éxito de los proyectos de IA en invasiones biológicas. Las
herramientas de aprendizaje profundo, como las CNN, destacan en su capacidad
para manejar datos visuales complejos, mientras que los modelos de ensamblaje
han mejorado la robustez de las predicciones al combinar múltiples algoritmos.
Sin embargo, persisten desafíos relacionados con la representatividad de los
datos, los sesgos inherentes y la opacidad de algunos algoritmos. Es crucial
priorizar el análisis crítico de los datos utilizados y fomentar el acceso a
bases de datos diversas y completas.
Las perspectivas futuras en el uso de la IA para especies
invasoras incluyen el desarrollo de modelos más integrados y adaptativos. La
combinación de enfoques mecanicistas y de aprendizaje profundo utilizando
aprendizaje por transferencia puede mejorar nuestra comprensión de los procesos
ecológicos subyacentes en base a conjuntos de datos más limitados y permitir
predicciones más precisas. Además, la democratización de las herramientas de IA
mediante plataformas accesibles y aplicaciones específicas fomentará la
colaboración interdisciplinaria y la integración de múltiples fuentes de datos.
Es esencial promover buenas prácticas, incluyendo la adherencia a principios
FAIR, evaluación de sesgos, la transparencia en los métodos y la evaluación
crítica de las herramientas utilizadas.
Contribución de los autores
EF: Conceptualización, curación de datos, análisis formal,
investigación, metodología, redacción – borrador original. RS-G: Supervisión,
Redacción – revisión y edición.
Disponibilidad de datos
La lista de artículos producto de la revisión sistemática y
el código para generar las figuras se encuentra disponible públicamente en el
GitHub de los autores: https://github.com/erolafr/AIForInvasionBiology_Ecosistemas/,
así como material complementario y traducciones al inglés.
Financiación, permisos requeridos, potenciales
conflictos de interés y agradecimientos
Los autores/as declaran no
tener ningún conflicto de intereses.
E.F. agradece la financiación
por parte de UKRI Marie Skłodowska-Curie Actions Fellowship (EP/Y02873/1) bajo
la supervisión de R.S.G. R.S.G. agradece la financiación de NERC Pushing the
Frontiers grant (NE/X013766/1). No hay conflictos de interés.
Referencias
Agathokleous, E., Saitanis, C
J., Fang, C., Yu, Z. 2023. Use of ChatGPT: What does it mean
for biology and environmental science? Science of the Total Environment
888: 164154. https://doi.org/10.1016/j.scitotenv.2023.164154
Adepoju, K.,
Adelabu, S. Mokubung, C. 2020. Mapping Seriphium
plumosum encroachment and investigating its drivers using Sentinel-2 and
environmental data. Environmental Monitoring and Assessment 192: 328. https://doi.org/10.1007/s10661-020-08253-x
Anandhavalli, M., Ghose, MK., Gauthaman, K. 2010. Association rule mining in
genomics. International Journal of Computer Theory and Engineering 2(2):
269-273. https://doi.org/10.7763/IJCTE.2010.V2.151
Araújo, M.B., Anderson, R.P., Barbosa, A.M., Beale, C.M., Dormann, C.F.,
Early, R., Garcia, R.A., et al. 2019. Standards for distribution models in
biodiversity assessments. Science Advances 5: eaat4858. https://doi.org/10.1126/sciadv.aat4858
Aussem, A.,
Hill, D. 1999. Wedding connectionist and
algorithmic modelling: applications to ecological systems. Ecological
Modelling 120(2-3): 225-236. https://doi.org/10.1016/S0304-3800(99)00104-0
Bacher, S., Ryan-Colton, E., Coiro, M., Cassey, P., Galil; B.S., Nuñez,
M.A., Ansong, M., et al. 2025. Global Impacts Dataset of Invasive Alien Species
(GIDIAS). Scientific Data 12, 832. https://doi.org/10.1038/s41597-025-05184-5
Bao, G., Weng, B., Xv, H. 2024. Application of deep neural network
identification models based on garden environment perception in plant
protection. Crop Protection 184: 106833. https://doi.org/10.1016/j.cropro.2024.106833
Barbosa, F.G., Schneck, F., Melo, A.S. 2012. Use of ecological niche models
to predict the distribution of invasive species: a scientometric analysis. Brazilian
Journal of Biology 72(4): 821-829. https://doi.org/10.1590/S1519-69842012000500007
Bednarsek, N., Beck, M.W., Pelletier, G., Applebaum, S.L., Feely, R.A.,
Butler, R., Byrne, M., et al. 2022. Natural analogues in pH variability and
predictability across the coastal pacific estuaries: extrapolation of the
increased oyster dissolution under increased pH amplitude and low
predictability related to ocean acidification. Environmental Science and
Technology 56(12): 9015-9028. https://doi.org/10.1021/acs.est.2c00010
Bae, M.-J., Kim, E.-J., Park, Y.-S. 2021. Comparison of invasive apple
snail (Pomacea canaliculata) behaviors in different water temperature
gradients. Water 13(9): 1149. https://doi.org/10.3390/w13091149
Ben-Hur, A., Ong, C.S., Sonnenburg, S., Schölkopf, B., Rätsch, G. 2008. Support vector machines and kernels for computational biology. PLoS
Computational Biology 4: e1000173. https://doi.org/10.1371/journal.pcbi.1000173
Benzer, S., Benzer, R. 2020. Growth and length-weight
relationships of Pseudorasbora parva (Temminck & Schlegel, 1846) in
Hirfanli Dam Lake: Comparison with traditional and artificial neural networks
approaches. Iranian Journal of Fisheries Sciences 19(3): 1098-1110.
Biswas, S.S. 2023. Potential use of chatGPT in global warming. Annals
of Biomedical Engineering 51: 1126-1127. https://doi.org/10.1007/s10439-023-03171-8
Blonder, B.W., Lim, M.H., Godoy, O. 2024. Predicting and prioritising
community assembly: learning outcomes via experiments. Ecology Letters
27(10): e14535. https://doi.org/10.1111/ele.14535
Bolaños, F., Salatino, A., Osborne, F., Motta, E. 2024. Artificial intelligence for literature reviews: opportunities and
challenges. Artificial Intelligence Review 57: 259. https://doi.org/10.1007/s10462-024-10902-3
Borowiec, M.L., Dikow, R.B., Frandsen, P.B., McKeeken, A., Valentini, G.,
White, A.E. 2022. Deep learning as a tool for ecology and evolution. Methods
in Ecology and Evolution 13: 1640-1660. https://doi.org/10.1111/2041-210X.13901
Bravo-Diaz, A., Moreno, S., Lopatin, J. 2024. Evaluating
the ability of convolutional neural networks for transfer learning in Pinus
radiata cover predictions. Ecological Informatics 82: 102684. https://doi.org/10.1016/j.ecoinf.2024.102684
Briscoe, N.J., Elith, J., Salguero‐Gómez, R., Lahoz‐Monfort,
J.J., Camac, J.S., Giljohann, K.M., Holden, M.H., et al. 2019. Forecasting species range dynamics
with process‐explicit models: matching methods to applications. Ecology Letters 22: 1940-1956. https://doi.org/10.1111/ele.13348
Buchelt, A., Adrowitzer, A., Kieseberg, P., Gollob, C., Nothdurft, A.,
Eresheim, S., Tschiatschek, S., et al. 2024. Exploring
artificial intelligence for applications of drones in forest ecology and
management. Forest Ecology and Management 551: 121530. https://doi.org/10.1016/j.foreco.2023.121530
Bzdok, D., Krzywinski, M., Altman, N. 2018. Machine learning: supervised
methods. Nature Methods 15: 5-6. https://doi.org/10.1038/nmeth.4551
Camacho, C.A., Sullivan, C.J.,
Weber, M.J., Pierce, C.L. 2019. Morphological identification
of bighead carp, silver carp, and grass carp eggs using random forests machine
learning classification. North American Journal of Fisheries Management
39: 1373-1384. https://doi.org/10.1002/nafm.10380
Cao, Y., Geddes, T.A., Yang, J.Y.H., Yang P. 2020. Ensemble deep
learning in bioinformatics. Nature Machine
Intelligence 2: 500-508. https://doi.org/10.1038/s42256-020-0217-y
Cardoso, A.S., Malta-Pinto, E., Tabik, S. 2024. Can
citizen science and social media images support biodiversity monitoring? Ecological
Informatics 81: 102602. https://doi.org/10.1016/j.ecoinf.2024.102602
Castillo, C., Chollett, I., Klein, E. 2008. Enhanced duckweed detection
using bootstrapped SVM classification on medium resolution RGB MODIS imagery. International
Journal of Remote Sensing 29(19): 5595-5604. https://doi.org/10.1080/01431160801961375
Cavender-Bares, J., Schneider, F.D., Santos, M.J., Amstrong, A., Carnaval, A,
Dahlin, K.M., Fatoyinbo, L., et al. 2022. Integrating remote sensing with
ecology and evolution to advance biodiversity conservation. Nature Ecology
and Evolution 6, 506–519. https://doi.org/10.1038/s41559-022-01702-5
Chaity, M.D.,
van Aardt, J. 2024. Exploring
the limits of species identification via a convolutional neural network in a
complex forest scene through simulated imaging spectroscopy. Remote Sensing 16(3): 498. https://doi.org/10.3390/rs16030498
Charlton, G., Falzon, G., Shepley, A., Fleming, P.J.S., Ballard, G., Meek,
P.D. 2024. The Sentinel Bait Station: an automated, intelligent design pest
animal baiting system. Wildlife Research 51(1). https://doi.org/10.1071/WR22183
Chen, G., Kéry, M., Plattner, M., Ma, K., Gardner, B. 2013. Imperfect
detection is the rule rather than the exception in plant distribution studies. Journal
of Ecology 101: 183-191. https://doi.org/10.1111/1365-2745.12021
Chen, Q., Xie, J., Yu, Q., Liu, C., Ding, W., Li, X., Zhou, H. 2024. An
experimental study of acoustic bird repellents for reducing bird encroachment
in pear orchards. Frontiers in Plant Science 15: 1365275. https://doi.org/10.3389/fpls.2024.1365275
Christin, S., Hervet, É., Lecomte, N. 2019. Applications for deep learning
in ecology. Methods in Ecology and Evolution 10: 1632-1644. https://doi.org/10.1111/2041-210X.13256
Clemmensen,
L., Kjærsgaard, R. 2022. Data representativity for
machine learning and AI systems. arXiv: 2203.04706v1.
Coughlan, N.E., Lyne, L., Cuthbert, R.N., Cunningham, E.M., Lucy, F., Davis,
E., Caffrey, J.M., Dick, J.T. 2020. In the black: Information harmonisation and
educational potential amongst international databases for invasive alien
species designated as of Union Concern. Global Ecology and Conservation
241. e01332. https://doi.org/10.1016/j.gecco.2020.e01332
Dar, S.A., Sharief, A., Kumar, V., Singh, H., Joshi, B.D.,
Bhattacharjee, S., Dutta, R., et al. 2023. Free-ranging dogs are seriously
threatening Himalayan environment: delineating the high-risk areas for curbing
free-ranging dog infestation in the Trans-Himalayan region. Environmental
Monitoring and Assessment 195: 1386. https://doi.org/10.1007/s10661-023-11972-6
Dara, R., Hazrati Fard, S.M., Kaur, J. 2022. Recommendations
for ethical and responsible use of artificial intelligence in digital
agriculture. Frontiers in Artificial Intelligence 5: 884192. https://doi.org/10.3389/frai.2022.884192
Dash, J.P., Watt, M.S., Paul, T.S.H., Morgenroth, J., Hartley, R. 2019.
Taking a closer look at invasive alien plant research: A review of the current
state, opportunities, and future directions for UAVs. Methods in Ecology and
Evolution 10: 2020-2033. https://doi.org/10.1111/2041-210X.13296
Davis, A.J.S., Groom, Q., Adriaens, T., Vanderhoeven, S., De Troch, R.,
Oldoni, D., Desmet, P., et al. 2024. Reproducible WiSDM: a workflow for
reproducible invasive alien species risk maps under climate change scenarios
using standardized open data. Frontiers in Ecology and Evolution 12:
1148895. https://doi.org/10.3389/fevo.2024.1148895
Deng, J., Dong, W., Socher, R., Li, L. -J., Li, K., Fei-Fei, L. 2009. ImageNet:
A large-scale hierarchical image database. IEEE Conference on Computer
Vision and Pattern Recognition, Miami, FL, USA, 248-255. https://doi.org/10.1109/CVPR.2009.5206848
Depauw, L., Blondeel, H., De
Lombaerde, E., De Pauw, K., Landuyt, D., Lorer, E., Vangansbeke, P., et al.
2022. The use of photos to investigate ecological change. Journal
of Ecology 110: 1220–1236. https://doi.org/10.1111/1365-2745.13876
Devi, N., Sarma, K.K., Laskar, S.
2023. Design of an intelligent bean cultivation approach using
computer vision, IoT and spatio-temporal deep learning structures. Ecological
Informatics 75: 102044. https://doi.org/10.1016/j.ecoinf.2023.102044
Diagne, C., Leroy, B., Gozlan Vaissière, A.-C., Assailly, C., Nuninger,
L., Roiz, D., et al. 2020. InvaCost, a public database of the economic costs of
biological invasions worldwide. Scientific data 7: 277. https://doi.org/10.1038/s41597-020-00586-z
Dietterich, T.G. 2000. Ensemble methods in machine learning. In: Multiple
classifier systems. MCS 2000. Lecture notes in computer science, vol 1857.
Springer, Berlin, Heidelberg. https://doi.org/10.1007/3-540-45014-9_1
Dyrmann, M., Skovsen, S.K., Christiansen, P.H., Kragh, M.F., Mortensen,
A.K. 2024. High-speed camera system for efficient monitoring of invasive plant
species along roadways. F1000Research 13: 360. https://doi.org/10.12688/f1000research.141992.2
Elith, J., Graham, C., NCEAS species distribution modeling group. 2006.
Novel methods improve prediction of species’ distributions from occurrence
data. Ecography 29: 129-151. https://doi.org/10.1111/j.2006.0906-7590.04596.x
Elith, J., Kearney, M., Phillips, S. 2011. The art of modelling
range-shifting species. Methods in Ecology and Evolution 1(4), 330–342. https://doi.org/10.1111/j.2041-210X.2010.00036.x
Epstein, J.M., Pine, W.E. III., Romagosa, C.M., Scott, M.C., Phillips,
C.T., Marion, C.A. Baiser, B. 2018. State- and regional-scale patterns and
drivers of freshwater fish functional diversity in the southeastern USA. Transactions
of the American Fisheries Society 147(6): 1179-1198. https://doi.org/10.1002/tafs.10110
Fenner, M. 1998. The phenology of growth and reproduction in plants. Perspectives
in Plant Ecology, Evolution and Systematics 1(1): 78-91. https://doi.org/10.1078/1433-8319-00053
Fenollosa, E., Pang, S.,
Briscoe, N., Guisan, A, Salguero-Gómez, R. 2025. Powerful yet
challenging: Mechanistic Niche Models for predicting invasive species potential
distribution under climate change. Ecography. https://doi.org/10.1002/ecog.07775
Fleure, V., Magneville, C., Mouillot, D. Villeger, S. 2024. Automated
identification of invasive rabbitfishes in underwater images from the
Mediterranean Sea. Aquatic Conservation: Marine and Freshwater Ecosystems 34(1).
https://doi.org/10.1002/aqc.4073
Fonnesbeck, C.J. 2008. Solving dynamic wildlife resource optimization problems
using reinforcement learning. Natural Resource Modeling 18(1): 1-40. H. https://doi.org/10.1111/j.1939-7445.2005.tb00147.x
Fourcade, Y. 2021. Fine-tuning niche models matters in invasion ecology: A
lesson from the land planarian Obama nungara. Ecological Modelling
457: 109686. https://doi.org/10.1016/j.ecolmodel.2021.109686
Galaz García, C., Bagstad, K.J., Brun, J., Chaplin-Kramer, R., Dhu, T., Murray,
N.J., Nolan, C.J., et al. 2023. The future of ecosystem assessments is
automation, collaboration, and artificial intelligence. Environmental Research Letters 18(1): 011003. https://doi.org/10.1088/1748-9326/acab19
García-Navarrete, O.L., Correa-Guimaraes, A., Navas-Gracia, L.M. 2024. Application of convolutional neural networks in weed detection and
identification: A systematic review. Agriculture 14: 568. https://doi.org/10.3390/agriculture14040568
Gil-Tapetado, D., Koken, M.,
De Cock, R., Gomez, J.F., Cabrero-Sanudo, F.J., Polidori, C. 2024. Across the firefly-verse: comparison of niche suitability of an
exotic firefly in its native and colonized range. Journal of Insect
Conservation 28(1): 43-56. https://doi.org/10.1007/s10841-023-00522-7
Gonzalez, L.F., Montes, G.A., Puig, E., Johnson, S., Mengersen, K., Gaston,
K.J. 2016. Unmanned aerial vehicles (UAVs) and artificial intelligence
revolutionizing wildlife monitoring and conservation. Sensors 16(1): 97.
https://doi.org/10.3390/s16010097
Goode, K., Weber, M.J., Dixon, P.M. 2023. WhoseEgg: classification
software for invasive carp eggs. PeerJ 11: e14787. https://doi.org/10.7717/peerj.14787
Greener, J.G., Kandathil, S.M., Moffat, L., Jonas,
D.T. 2022. A guide to machine learning for biologists. Nature
Reviews Molecular Cell Biology 23: 40-55. https://doi.org/10.1038/s41580-021-00407-0
Gugele, S.M., Widmer, M., Baer, J., DeWeber, J.T., Balk, H., Brinker, A.
2021. Differentiation of two swim bladdered fish
species using next generation wideband hydroacoustics. Scientific Reports 11(1):
10520. https://doi.org/10.1038/s41598-021-89941-7
Guisan, A., Petitpierre, B., Broennimann, O., Daehler, C., Kueffer, C.
2014. Unifying niche shift studies: insights from biological invasions. Trends
in Ecology and Evolution 29: 260-269. https://doi.org/10.1016/j.tree.2014.02.009
Guisan, A., Thuiller, W., Zimmermann, N.E. 2017. Habitat suitability
and distribution models: with applications in R. Cambridge University
Press. https://doi.org/10.1017/9781139028271
Gupta, K., Rani, R., Bahia, N. 2020. Plant-seedling
classification using transfer learning. International Journal of
Agricultural and Environmental Information Systems 11(4): 25-40. https://doi.org/10.4018/IJAEIS.2020100102
Haase, C.G., Yang, A., McNyset, K.M., Blackburn, J.K. 2021. GARPTools: R
software for data preparation and model evaluation of GARP models. Ecography
44: 1790-1796. https://doi.org/10.1111/ecog.05642
Hamilton,
S.A., Murphy, D.D. 2020. Use of affinity analysis
to guide habitat restoration and enhancement for the imperiled delta smelt.
Endangered Species Research 43:103-120. https://doi.org/10.3354/esr01057
Han, B.A., Varshney, K. R., LaDeau, S., Subramaniam, A., Weathers,
K.C., Zwart, J. 2023. A synergistic future for AI and ecology. Proceedings
of the National Academy of Sciences of the United States of America
120(38): e2220283120. https://doi.org/10.1073/pnas.2220283120
Hastie, T., Tibshirani, R., Friedman, J. 2009. The
elements of statistical learning: data mining, inference, and prediction. 2ª ed. Springer Science and Business Media.
Heger, T., Jeschke, J.M. Kollmann, J. 2021. Some reflections on current
invasion science and perspectives for an exciting future. NeoBiota 68:
79-100. https://doi.org/10.3897/neobiota.68.68997
Heil, B.J., Hoffman, M.M., Markowetz, F., Lee, S.-I., Greene, C.S., Hicks,
S.C. 2021. Reproducibility standards for machine
learning in the life sciences. Nature Methods 18: 1132-1135. https://doi.org/10.1038/s41592-021-01256-7
Hodgson, J.C., Mott, R., Baylis, S.M., Pham, T.T., Wotherspoon, S.,
Kilpatrick, A.D., Segaran, R.R., et al. 2018. Drones count wildlife more
accurately and precisely than humans. Methods in Ecology and Evolution
9: 1160–1167. https://doi.org/10.1111/2041-210X.12974
Hollings, T., Burgman, M., van
Andel, M., Gilbert, M., Robinson, T., Robinson, A. 2018. How
do you find the green sheep? A critical review of the use of remotely sensed
imagery to detect and count animals. Methods in Ecology and Evolution 9:
881-892. https://doi.org/10.1111/2041-210X.12973
Hui, C. 2023. The dos and don’ts for predicting invasion dynamics with
species distribution models. Biological Invasions 25: 947-953. https://doi.org/10.1007/s10530-022-02976-3
Iguchi, K., Matsuura, K., McNyset, K.M., Peterson, A.T.,
Scachetti-Pereira, R., Powers, K.A., Vieglais, D.A., et al. 2004. Predicting
invasions of North American basses in Japan using native range data and a
genetic algorithm. Transactions of the American Fisheries Society
133(4): 845-854. https://doi.org/10.1577/T03-172.1
Jain, A.K. 2010. Data clustering: 50 years beyond K-means. Pattern
Recognition Letters 31: 651–666. https://doi.org/10.1016/j.patrec.2009.09.011
James, K.
Bradshaw, K. 2020. Detecting
plant species in the field with deep learning and drone technology. Methods
in Ecology and Evolution 11(11): 1509-1519. https://doi.org/10.1111/2041-210X.13473
Javed, Q., Azeem, A., Sun, J., Ullah, I., Du, D., Imran, M.A., Nawaz,
M.I., Chattha, H.T. 2022. Growth prediction of Alternanthera philoxeroides
under salt stress by application of artificial neural networking. Plant
Biosystems 156(1): 61-67. https://doi.org/10.1080/11263504.2020.1832603
Jeschke,
J.M., Strayer, D.L. 2008. Usefulness of bioclimatic
models for studying climate change and invasive species. En: Ostfeld, R.S.,
Schlesinger, W.H. (eds.). Year in Ecology and Conservation Biology 2008. Annals
of the New York Academy of Sciences 1134: 1-24. https://doi.org/10.1196/annals.1439.002
Jia,
H., Cai, D., Yang, J., Qian, W., Wang, C. 2023. Efficient
and privacy-preserving image classification using homomorphic encryption and
chunk-based convolutional neural network. Journal of Cloud Computing 12:
175. https://doi.org/10.1186/s13677-023-00537-0
Jiang, D., Chen, S., Hao, M., Fu, J., Ding, F. 2018. Mapping the
potential global codling moth (Cydia pomonella L.) distribution based on
a machine learning method. Scientific Reports 8: 13093. https://doi.org/10.1038/s41598-018-31478-3
Katal, N., Rzanny, M., Mäder, P., Wäldchen, J. 2022. Deep learning in
plant phenological research: A systematic literature review. Frontiers in
Plant Science 13: 805738. https://doi.org/10.3389/fpls.2022.805738
Kattenborn, T., Eichel, J., Wiser, S.,
Burrows, L., Fassnacht, F.E., Schmidtlein, S. 2020. Convolutional
neural networks accurately predict cover fractions of plant species and
communities in unmanned aerial vehicle imagery. Remote Sensing in Ecology
and Conservation 6(4): 472-486. https://doi.org/10.1002/rse2.146
Kattge, J., Bönisch, G., Díaz, S., Lavorel, S., Colin Prentice, I., Leadley,
P., Tautenhahn, S., et al. 2020. TRY plant trait
database – enhanced coverage and open access. Global Change Biology 26:
119-188. https://doi.org/10.5194/egusphere-egu2020-20191
Keerthinathan, P.,
Amarasingam, N., Desilva, A. 2024. African lovegrass
segmentation with artificial intelligence. Remote Sensing 16(13): 2363. https://doi.org/10.3390/rs16132363
Keller, R.P., Kocev, D., Dzeroski, S. 2011. Trait-based risk assessment
for invasive species: high performance across diverse taxonomic groups,
geographic ranges and machine learning/statistical tools. Diversity and
Distributions 17(3): 451-461. https://doi.org/10.1111/j.1472-4642.2011.00748.x
Knight, E., Hannah, K., Foley, G., Scott, C., Brigham, R., Bayne, E. 2017.
Recommendations for acoustic recognizer performance assessment with application
to five common automated signal recognition programs. Avian Conservation and
Ecology 12(2). https://doi.org/10.5751/ACE-01114-120214
Kolar, C. 2004. Risk assessment and screening for potentially invasive
fishes. New Zealand Journal of Marine and Freshwater Research 38(3):
391-397. https://doi.org/10.1080/00288330.2004.9517247
Kotowska, D., Pärt, T., Żmihorski, M. 2021. Evaluating Google Street View
for tracking invasive alien plants along roads. Ecological Indicators
121: 107020. https://doi.org/10.1016/j.ecolind.2020.107020
Krizhevsky, A., Sutskever, I., Hinton, G. 2017. ImageNet classification with
deep convolutional neural networks. Communications of the ACM 60(6):
84-90. https://doi.org/10.1145/3065386
Kulkarni,
R., Di Minin, E. 2023. Towards automatic detection
of wildlife trade using machine vision models. Biological Conservation
279: 109924. https://doi.org/10.1016/j.biocon.2023.109924
Lapeyrolerie, M., Chapman, M.S., Norman, K.E.A., Boettiger, C. 2022. Deep
reinforcement learning for conservation decisions. Methods in Ecology and
Evolution 13: 2649–2662. https://doi.org/10.1111/2041-210X.13954
Latombe, G., Pyšek, P., Jeschke, J.M., Blackburn, T.M., Bacher, S.,
Capinha, C., Costello, M.J., et al. 2017. A vision for global monitoring of
biological invasions. Biological Conservation 213(B): 295-308. https://doi.org/10.1016/j.biocon.2016.06.013
Leathwick, J.R., Elith, J., Hastie, T. 2006. Comparative performance of
generalized additive models and multivariate adaptive regression splines for
statistical modelling of species distributions. Ecological Modelling
199: 188-196. https://doi.org/10.1016/j.ecolmodel.2006.05.022
Lehman, A., Overton, J.M., Leathwick, J.R. 2002. GRASP: generalized
regression analysis and spatial prediction. Ecological
Modelling 157: 189-207. https://doi.org/10.1016/S0304-3800(02)00195-3
Lenzner, B., Latombe, G., Schertler, A., Seebens, H., Yang, Q., Winter, M.,
Weigelt, P., et al. 2022. Naturalized alien floras
still carry the legacy of European colonialism. Nature Ecology and Evolution
6(11), 1723–1732. https://doi.org/10.1038/s41559-022-01865-1
Levin, S.C., Evers, S., Potter, T., Guerrero, M. P., Childs, D.Z.,
Compagnoni, A., Knight, T.M., Salguero-Gómez, R. 2022. Rpadrino: An R package
to access and use PADRINO, an open access database of Integral Projection
Models. Methods in Ecology and Evolution 13: 1923-1929. https://doi.org/10.1111/2041-210X.13910
Ley, C., Martin, R.K., Pareek, A., et al. 2022. Machine learning and
conventional statistics: making sense of the differences. Knee Surgery,
Sports Traumatology, Arthroscopy 30: 753-757. https://doi.org/10.1007/s00167-022-06896-6
Li, C., Bo, H., Song, B., Chen, X., Cao, Q., Yang, R., Ji, S., et al.
2022. Reshaping of the soil microbiome by the expansion of invasive plants:
shifts in structure, diversity, co-occurrence, niche breadth, and assembly
processes. Plant and Soil 477: 629-646. https://doi.org/10.1007/s11104-022-05445-x
Los, S., Mücher, C.A., Kramer, H., Franke, G.J., Kamphuis, C. 2023.
Estimating body dimensions and weight of cattle on pasture with 3D models from
UAV imagery. Smart Agricultural Technology 4: 100167. https://doi.org/10.1016/j.atech.2022.100167
Loyani, L. 2024. Segmentation-based quantification of Tuta absoluta's
damage on tomato plants. Smart Agricultural Technology 7: 100415. https://doi.org/10.1016/j.atech.2024.100415
Ma, D., Wei, J., Zhu, L., Zhao, F., Wu, H., Chen, X., Li, Y., et al.
2024. Semi-supervised learning advances species recognition for aquatic
biodiversity monitoring. Frontiers in Marine Science 11: 1373755. https://doi.org/10.3389/fmars.2024.1373755
Maher, J., Stringham, O.C., Moncayo, S., Wood, L., Lassaline, C.R.,
Virtue, J., Cassey, P. 2023. Weed wide web: characterising illegal online trade
of invasive plants in Australia. NeoBiota 87: 45-72. https://doi.org/10.3897/neobiota.87.104472
Maitner, B.S., Boyle, B., Casler,
N., Condit, R., Donoghue II, J., Durán, S.M., Guaderrama, D., et al. 2018. The bien r package: A tool to access the Botanical
Information and Ecology Network (BIEN) Database. Methods in Ecology and
Evolution 9: 373–379. https://doi.org/10.1111/2041-210X.12861
Mammola, S.,
Leroy, B. 2018. Applying species distribution
models to caves and other subterranean habitats. Ecography 41: 1194-1208.
https://doi.org/10.1111/ecog.03464
Marshall, D.J. 2024. Principles of experimental design for ecology and
evolution. Ecology Letters 27: e14400. https://doi.org/10.1111/ele.14400
Martinez, B., Reaser, J.K., Dehgan, A., Zamft, B., Baisch, D., McCormick,
C., Giordano, A.J., et al. 2020. Technology innovation: advancing capacities
for the early detection of and rapid response to invasive species. Biological Invasions 22: 75-100. https://doi.org/10.1007/s10530-019-02146-y
Martínez-Vega, J., Diaz, A., Miguel
Nava, J., Gallardo, M., Echavarria, P. 2017. Assessing
land use-cover changes and modelling change scenarios in two mountain Spanish
national parks. Environments 4(4): 79. https://doi.org/10.3390/environments4040079
Marzialetti, F., Frate, L., De
Simone, W., Frattaroli, A.R., Acosta, A.T.R., Carranza, M.L. 2021. Unmanned aerial vehicle (UAV)-based mapping of Acacia saligna
invasion in the mediterranean coast. Remote Sensing 13(17): 3361. https://doi.org/10.3390/rs13173361
Mathur, M.,
Mathur, P. 2024. Habitat suitability of Opuntia
ficus-indica (L.) Mill. (Cactaceae): a comparative temporal evaluation
using diverse bio-climatic earth system models and ensemble machine learning
approach. Environmental Monitoring and Assessment 196(3): 232. https://doi.org/10.1007/s10661-024-12406-7
Matsuhashi, S., Oishi, Y., Koarai, A., Sugiura, R. 2024. Enhancing image
recognition robustness in early weed detection through optimal training data
curation. Weed Science 72(5):546-552. https://doi.org/10.1017/wsc.2024.63
Meerdink, S., Hiatt, D., Flory, S.L., Zare, A. 2024. Dealing with imperfect
data for invasive species detection using multispectral imagery. Ecological
Informatics 79: 102432. https://doi.org/10.1016/j.ecoinf.2023.102432
Morey, A.C.,
Venette, R.C. 2020. Minimizing risk and maximizing
spatial transferability: challenges in constructing a useful model of potential
suitability for an invasive insect. Annals of the Entomological Society of
America 113(2): 100-113. https://doi.org/10.1093/aesa/saz049
Mqingwana, P., Shoko, C., Gxokwe, S., Dube, T. 2024. Monitoring and assessing
the effectiveness of the biological control implemented to address the invasion
of water hyacinth (Eichhornia crassipes) in Hartbeespoort Dam, South
Africa. Remote Sensing Applications: Society and Environment 36: 101295.
https://doi.org/10.1016/j.rsase.2024.101295
Nazir, S., Kaleem, M. 2021. Advances in image acquisition and
processing technologies transforming animal ecological studies. Ecological
Informatics 61, 101212. https://doi.org/10.1016/j.ecoinf.2021.101212
Nisin, M.K.M.N., Ramanathan, S.K.K., Sreeram, M.P., Sudheesan, D. 2023.
Ensemble modelling of Pirarucu (Arapaima gigas) distribution in
biodiversity hotspot to understand its invasion risk. Ecology of Freshwater
Fish 32(3): 528-537. https://doi.org/10.1111/eff.12704
Novoa, A., Jarić, I., Pipek, P., Pyšek, P. 2025.
Culturomics and iEcology provide novel opportunities to study human and social
dimensions of alien species introductions. Trends in Ecology and Evolution
40 (1), 18-26. https://doi.org/10.1016/j.tree.2024.08.012
Nunez-Mir, G.C., Iannone, B.V. III., Curtis, K., Fei, S. 2015. Evaluating the evolution of forest restoration research in a
changing world: a big literature review. New Forests 46(5-6): 669-682. https://doi.org/10.1007/s11056-015-9503-7
Olden, J.D., Lawler, J.J., Poff, N.L. 2008. Machine learning methods
without tears: A primer for ecologists. The Quarterly Review of Biology 83(2):
171-193. https://doi.org/10.1086/587826
Olivoto, T. 2022. Lights, camera, pliman! An R package for plant image
analysis. Methods in Ecology and Evolution 13: 789-798. https://doi.org/10.1111/2041-210X.13803
Orenstein, E.C., Ayata, S.-D., Maps, F., Becker, É.C., Benedetti, F., Biard,
T., de Garidel-Thoron, T., et al. 2022. Machine learning techniques to
characterize functional traits of plankton from image data. Limnology and
Oceanography 67: 1647-1669. https://doi.org/10.1002/lno.12101
Orouji, S., Liu, M.C., Korem, T., Peters, M.A.K. 2024. Domain adaptation
in small-scale and heterogeneous biological datasets. Science Advances
10(51). https://doi.org/10.1126/sciadv.adp6040
O'Shea-Wheller, T.A., Corbett, A., Osborne, J.L., Recker, M., Kennedy, P.J. 2024.
VespAI: a deep learning-based system for the detection of invasive hornets. Communications
Biology 7(1). https://doi.org/10.1038/s42003-024-05979-z
Pagad, S., Genovesi, P., Carnevali, L., Schigel, D., McGeoch, M.A. 2018. Introducing the Global Register of Introduced and Invasive Species.
Scientific data 5: 170202. https://doi.org/10.1038/sdata.2017.202
Palazzetti, L., Rangarajan, A.K., Dinca, A., Boom, B., Popescu, D., Offermans,
P., Pinotti, C.M. 2024. The hawk eye scan: Halyomorpha halys detection
relying on aerial tele photos and neural networks. Computers and Electronics
in Agriculture 226: 109365. https://doi.org/10.1016/j.compag.2024.109365
Pan, S.J., Yang, Q. 2010. A survey on transfer learning.
IEEE Transactions on Knowledge and Data Engineering 22: 1345-1359. https://doi.org/10.1109/TKDE.2009.191
Pasha, S.V.,
Reddy, C.S. 2024. Global
spatial distribution of Prosopis juliflora - one of the world's worst
100 invasive alien species under changing climate using multiple machine
learning models. Environmental Monitoring and Assessment 196(2): 196. https://doi.org/10.1007/s10661-024-12347-1
Patel, M., Jernigan, S., Richardson, R., Ferguson, S., Buckner, G. 2019.
Autonomous robotics for identification and management of invasive aquatic plant
species. Applied Sciences 9(12):2410. https://doi.org/10.3390/app9122410
Pearson, R.G. 2003. Species’ distribution modeling for conservation
educators and practitioners. Lessons in conservation 3., 54-89: Network of
conservation educators and practitioners, Center for Biodiversity and
Conservation, American Museum of Natural History. https://doi.org/10.5531/cbc.linc.3.1.3
Pearson, R.G., Dawson, T.P., Berry, P.M., Harrison, P.A. 2002. Species: A
spatial evaluation of climate impact on the envelope of species. Ecological
Modelling 154: 289-300. https://doi.org/10.1016/S0304-3800(02)00056-X
Philibert, A., Desprez-Loustau, M.L., Fabre, B., Frey, P., Halkett, F.,
Husson, C., Lung-Escarmant, B., et al. 2011. Predicting invasion success of
forest pathogenic fungi from species traits. Journal of Applied Ecology
48(6): 1381-1390. https://doi.org/10.1111/j.1365-2664.2011.02039.x
Phillips, S.J., Anderson, R.P., Schapire, R.E. 2006. Maximum entropy
modeling of species geographic distributions. Ecological Modelling 190:
231-259. https://doi.org/10.1016/j.ecolmodel.2005.03.026
Pichler, M.,
Hartig, F. 2023. Machine learning and deep
learning—a review for ecologists. Methods in Ecology and Evolution
14(4): 994-1016. https://doi.org/10.1111/2041-210X.14061
Polverino, G., Soman, V.R., Karakaya, M., Gasparini, C., Evans, J.P.,
Porfiri, M. 2022. Ecology of fear in highly invasive fish revealed by robots.
iScience 25: 103529. https://doi.org/10.1016/j.isci.2021.103529
Pontin, D.R., Schliebs, S., Worner, S.P., Watts, M.J. 2011. Determining factors that influence the dispersal of a pelagic
species: A comparison between artificial neural networks and evolutionary
algorithms. Ecological Modelling 222(10): 1657-1665. https://doi.org/10.1016/j.ecolmodel.2011.03.002
Potapov, A. 2009. Stochastic model of lake system invasion and its optimal
control: neurodynamic programming as a solution method. Natural Resource Modeling 22(2):
257-288. https://doi.org/10.1111/j.1939-7445.2008.00036.x
Prado Osco, L., Wu, Q., Lopes de Lemos, E., Nunes Gonçalves, W., Marques
Ramos, A.P., Li, J., Marcato Junior, J. 2023. The
Segment Anything Model (SAM) for remote sensing applications: From zero to one
shot. International Journal of Applied Earth Observation and Geoinformation
124: 103540. https://doi.org/10.1016/j.jag.2023.103540
Prober, S.M., Hilbert, D.W., Ferrier, S., Dunlop, M., Gobbett, D. 2012.
Combining community-level spatial modelling and expert knowledge to inform
climate adaptation in temperate grassy eucalypt woodlands and related
grasslands. Biodiversity and Conservation 21(7): 1627-1650. https://doi.org/10.1007/s10531-012-0268-4
Pyron, R.A. 2023. Unsupervised machine learning for species delimitation,
integrative taxonomy, and biodiversity conservation. Molecular
Phylogenetics and Evolution 189: 107939. https://doi.org/10.1016/j.ympev.2023.107939
Raab, C., Stroh, H.G., Tonn, B., Meissner, M., Rohwer, N., Balkenhol, N., e
Isselstein, J. 2018. Mapping semi-natural grassland
communities using multi-temporal RapidEye remote sensing data. International
Journal of Remote Sensing 39(17): 5638-5659. https://doi.org/10.1080/01431161.2018.1504344
Rainio, O., Teuho, J., Klén, R. 2024. Evaluation metrics and statistical
tests for machine learning. Scientific Reports 14: 6086. https://doi.org/10.1038/s41598-024-56706-x
Rodas-Trejo, J.,
Ocampo-González, P. 2024. Assessment of ChatGPT's
potential as an innovative tool in searching for information on wild mammals. Ecological
Informatics 102810. https://doi.org/10.1016/j.ecoinf.2024.102810
Roberts,
J., Florentine, S. 2024. Advancements and
developments in the detection and control of invasive weeds: A global review of
the current challenges and future opportunities. Weed Science 72(3):
205-215. https://doi.org/10.1017/wsc.2024.13
Roy, H.E., Pauchard, A., Stoett, P.J., Renard Truong, T., Meyerson, L.A.,
Bacher, S., Galil, B.S., et al. 2024. Curbing the major and growing threats
from invasive alien species is urgent and achievable. Nature Ecology and
Evolution 8: 1216-1223.
Saffer, A., Worm, T., Takeuchi, Y., Meentemeyer, R. 2024. GIATAR: a
spatio-temporal dataset of global invasive and alien species and their traits. Scientific Data 11: 991. https://doi.org/10.1038/s41597-024-03824-w
Salcedo, E., Uchani, Y., Mamani, M., Fernandez, M. 2024. Towards continuous floating invasive plant removal using unmanned
surface vehicles and computer vision. IEEE Access 12: 6649-6662. https://doi.org/10.1109/ACCESS.2024.3351764
Salguero-Gómez,
R., Gamelon, M. (eds). 2021. Demographic methods
across the tree of life. Oxford University Press. Oxford. UK. https://doi.org/10.1093/oso/9780198838609.001.0001
Salguero-Gómez, R., Jones, O.R., Archer, C.R., Buckley, Y.M., Che-Castaldo, J.,
Caswell, H., Hodgson, D., et al. 2015. The compadre Plant Matrix Database: an
open online repository for plant demography. Journal
of Ecology 103: 202-218.
Salguero-Gómez, R., Jones, O.R., Archer, C.R., Bein, C., de Buhr, H., Farack, C.,
Gottschalk, F., et al. 2016. COMADRE: a global data base of animal demography. Journal
of Animal Ecology 85: 371-384. https://doi.org/10.1111/1365-2656.12482
Sampaio Franco, A.C., Garcia-Berthou, E., Neves dos Santos, L. 2021. Ecological impacts of an invasive top predator fish across South
America. Science of the Total Environment 761: 143296. https://doi.org/10.1016/j.scitotenv.2020.143296
Santoianni, L.A., Innangi, M.,
Varricchione, M., Carboni, M., La Bella, G., Haider, S., Stanisci, A. 2024. Ecological features facilitating spread of alien plants along
Mediterranean mountain roads. Biological Invasions 26(11): 3879-3899. https://doi.org/10.1007/s10530-024-03418-y
Schmickl, T.,
Romano, D. 2024. Robots and animals teaming up in
the wild to tackle ecosystem challenges. Science Robotics 9:
eado5566. https://doi.org/10.1126/scirobotics.ado5566
Sillero, N., Arenas-Castro, S.,
Enriquez-Urzelai, U., Vale, C.G., Sousa-Guedes, D., Martínez-Freiría, F., Real,
R., Mácia, B.A. 2021. Want to model a species niche? A
step-by-step guideline on correlative ecological niche modelling. Ecological
Modelling 456: 109671. https://doi.org/10.1016/j.ecolmodel.2021.109671
Silvestro, D., Goria, S., Sterner, T., Antonelli, A. 2022. Improving
biodiversity protection through artificial intelligence. Nature Sustainability 5(5): 415-424. https://doi.org/10.1038/s41893-022-00851-6
Simon, S.M., Glaum, P., Valdovinos, F.S. 2023. Interpreting random forest
analysis of ecological models to move from prediction to explanation. Scientific
Reports 13: 3881. https://doi.org/10.1038/s41598-023-30313-8
Singh, K.K., Surasinghe, T.D., Frazier, A.E. 2024. Systematic review and
best practices for drone remote sensing of invasive plants. Methods in
Ecology and Evolution 15: 998-1015. https://doi.org/10.1111/2041-210X.14330
Soley-Guardia, M., Alvarado-Serrano, D.F., Anderson, R.P. 2024. Top ten hazards
to avoid when modeling species distributions: a didactic guide of assumptions,
problems, and recommendations. Ecography 2024: e06852. https://doi.org/10.1111/ecog.06852
Stockwell, D., Peters, D. 1999. The GARP modelling system: problems and
solutions to automated spatial prediction. International Journal of
Geographical Information Science 13: 143-158. https://doi.org/10.1080/136588199241391
Stone, T.C.,
Davis, K.J. 2024. Using unmanned aerial vehicles to
estimate body volume at scale for ecological monitoring. Methods in Ecology
and Evolution 00: 1-15. https://doi.org/10.1101/2023.11.23.567408
Sudholz, A., Denman, S., Pople, A., Brennan, M., Amos, M., Hamilton, G.
2022. A comparison of manual and automated detection of rusa deer (Rusa
timorensis) from RPAS-derived thermal imagery. Wildlife Research
49(1): 46-53. https://doi.org/10.1071/WR20169
Toussaint, A., Beauchard, O., Oberdorff, T., Brosse, S., Villeger, S. 2014.
Historical assemblage distinctiveness and the introduction of widespread
non-native species explain worldwide changes in freshwater fish taxonomic
dissimilarity. Global Ecology and Biogeography 23(5): 574-584. https://doi.org/10.1111/geb.12141
Trisos, C.H., Auerbach, J., Katti, M. 2021. Decoloniality and
anti-oppressive practices for a more ethical ecology. Nature Ecology and
Evolution 5: 1205-1212. https://doi.org/10.1038/s41559-021-01460-w
Tsamardinos, I., Greasidou, E., Borboudakis, G. 2018. Bootstrapping the
out-of-sample predictions for efficient and accurate cross-validation.
Machine Learning 107: 1895-1922. https://doi.org/10.1007/s10994-018-5714-4
Ulhaq, A., Adams, P., Cox, T.E., Khan, A., Low, T., Paul, M. 2021.
Automated detection of animals in low-resolution airborne thermal imagery.
Remote Sensing 13(16): 3276. https://doi.org/10.3390/rs13163276
Urza, A.K., Board, D.I., Bradford, J.B., Brown, J.L., Chambers, J.C.,
Schlaepfer, D.R., Short, K.C. 2024. Disentangling drivers of annual grass
invasion: Abiotic susceptibility vs. fire-induced conversion to cheatgrass
dominance in the sagebrush biome. Biological Conservation 297: 110737. https://doi.org/10.1016/j.biocon.2024.110737
Van Belleghem, S.M., Papa, R., Ortiz-Zuazaga, H., Hendrickx, F., Jiggins, C.D.,
McMillan, W.O., Counterman, B.A., et al. 2018. patternize: An R package
for quantifying colour pattern variation. Methods in Ecology and Evolution
9: 390-398. https://doi.org/10.1111/2041-210X.12853
van Kleunen, M., Pyšek, W., Dawson, F., Essl, H., Kreft, J., Pergl, P.,
Weigelt, A., et al. 2019. The Global Naturalized Alien Flora (GloNAF)
database. Ecology 100(1):e02542.
Veiga Branco, V., Correia, L.,
Cardoso, P. 2023. The use of machine learning in species
threats and conservation analysis. Biological Conservation 283: 110091. https://doi.org/10.1016/j.biocon.2023.110091
Wacquet, G.,
Lefebvre, A. 2022. EcoTransLearn: an
R-package to easily use transfer learning for ecological studies—a plankton
case study. Bioinformatics 38(24): 5469-5471. https://doi.org/10.1093/bioinformatics/btac703
Walsh, I., Fishman, D., Garcia-Gasulla, D., Titma, T., Pollastri, G.,
ELIXIR Machine Learning Focus Group, Harrow, J., et al. 2021. DOME:
recommendations for supervised machine learning validation in biology. Nature
Methods 18: 1122-1127. https://doi.org/10.1038/s41592-021-01205-4
Wang, K., Franklin, S.E., Guo, X., Cattet, M. 2010. Remote sensing of ecology, biodiversity and conservation: a review
from the perspective of remote sensing specialists. Sensors
10, 9647–9667. https://doi.org/10.3390/s101109647
Wang, L., Silvan-Cardenas, J.L., Yang, J. Frazier, A.E. 2013. Invasive saltcedar (Tamarisk spp.) distribution mapping using
multiresolution remote sensing imagery. Professional Geographer 65(1):
1-15. https://doi.org/10.1080/00330124.2012.679440
Weller, H.I., Hiller, A.E., Lord, N.P., Van Belleghem, S.M. 2024. recolorize:
An R package for flexible colour segmentation of biological images. Ecology
Letters 27: e14378. https://doi.org/10.1111/ele.14378
White, D.T., Antoniou, T.M., Martin, J.M., Kmetz, W., Twiss, M.R. 2022. A
machine-learning approach to predict success of a biocontrol for invasive Eurasian
watermilfoil reduction. Ecological Applications 32(6): e2625. https://doi.org/10.1002/eap.2625
Wiegleb, J., Hirsch, P.E., Seidel, F., Rauter, G. Burkhardt-Holm, P. 2022.
Flow, force, behaviour: assessment of a prototype hydraulic barrier for
invasive fish. Hydrobiologia 849(4): 1001-1019. https://doi.org/10.1007/s10750-021-04762-z
Wilson, N. 2024. Artificial intelligence helps drive new frontiers in
ecology. BioScience 74(5): 306-311. https://doi.org/10.1093/biosci/biae016
Wood, C.M., Guenther, F., Rex, A., Hofstadter, D.F., Reers, H., Kahl,
S., Peery, M.Z., et al. 2024. Real-time acoustic monitoring facilitates the
proactive management of biological invasions. Biological Invasions 26(12):3989-3996. https://doi.org/10.1007/s10530-024-03426-y
Xiao, Y., Greiner, R. Lewis, M.A. 2018. Evaluation of machine learning
methods for predicting eradication of aquatic invasive species. Biological Invasions 20(9): 2485-2503. https://doi.org/10.1007/s10530-018-1730-3
Xiong, B., Han, S., Messerschmidt, T.C., Kirwan, M.L., Gedan, K., Qi, M.
2024. Early detection of invasive Phragmites
australis at the tidal marsh-forest ecotone with airborne LiDAR. Ecological Indicators 167: 112651. https://doi.org/10.1016/j.ecolind.2024.112651
Yang, L., Yan, J., Li, H., Cao, X., Ge, B., Qi, Z., Yan, X. 2022. Real-time classification of invasive plant seeds based on improved
YOLOv5 with attention mechanism. Diversity 14: 254. https://doi.org/10.3390/d14040254
Yang, W., Wan, X. Deng, M. 2024. Spatial ensemble learning for
predicting the potential geographical distribution of invasive species. International
Journal of Geographical Information Science 38(11): 2216-2234. https://doi.org/10.1080/13658816.2024.2376325
Zhang, M., Shi, C., Li, X., Wang, K., Qiu, Z., Shi, F. 2023. Changes in
the structure and function of rhizosphere soil microbial communities induced by
Amaranthus palmeri invasion. Frontiers in Microbiology 14:
1114388. https://doi.org/10.3389/fmicb.2023.1114388
Zhao, J.Q., Gao, T., Du, J.J., Shi, J. 2023a. Future trends in Obolodiplosis
robiniae distribution across the Eurasian continent under global climate
change. Insects 14(1): 48. https://doi.org/10.3390/insects14010048
Zhao, Z., Fan, C., Liu, L. 2023b. Geo SAM: A QGIS plugin using Segment
Anything Model (SAM) to accelerate geospatial image segmentation (1.1.0). Zenodo.
Zhu,
W., Ding, Z., Pan, Y., Wang, Q. 2022. Using an affinity
analysis to identify phytoplankton associations. Ecology and Evolution 12,
e9047. https://doi.org/10.1002/ece3.9047
Zhuang, J., Jin, X., Chen, Y., Meng, W., Wang, Y., Yu, J., Muthukumar, B.
2023. Drought stress impact on the performance of deep convolutional neural
networks for weed detection in bahiagrass. Grass and Forage Science 78(1):
214-223. https://doi.org/10.1111/gfs.12583
Zurell, D., Franklin, J., König, C., Bouchet, P.J., Dormann, C.F., Elith,
J., Fandos, G., et al. 2020. A standard protocol for reporting species
distribution models. Ecography 43: 1261-1277. https://doi.org/10.1111/ecog.04960
![]() , Roberto
Salguero-Gómez1
, Roberto
Salguero-Gómez1 ![]()



