El auge de la ciencia de datos en los últimos años
La ciencia de datos se ha convertido en una disciplina
crucial en numerosos campos de estudio, incluida la ecología (King 2011; Michener y
Jones 2012; Ellwood et al. 2020). Mediante
el empleo de tecnologías de la información, métodos estadísticos y herramientas
de visualización, se pueden identificar patrones y extraer información útil a
partir de datos en “bruto” (Michener y Jones 2012;
Wickham et al. 2023). La creciente cantidad de
información disponible en las últimas décadas ha generado la necesidad de
implementar nuevos softwares que faciliten el trabajo con grandes bases de
datos a usuarios no especializados en programación informática, como ocurre en
el campo de la ecología (Hampton et al. 2013; Farley et al. 2018). Estos softwares deben ser
intuitivos, de libre acceso y gratuitos para garantizar una accesibilidad
universal. Además, es fundamental que proporcionen entornos de trabajo que
fomenten la generación continua de material didáctico de consulta (webs, foros,
tutoriales, etc.) y la reproducibilidad del trabajo realizado (Fanelli 2018).
Existen varios lenguajes de programación populares,
gratuitos y de código abierto que se utilizan en ciencia de datos, incluyendo R
(R Core Team 2023), Python (Van Rossum y Drake 1995) o Julia (Bezanson et al. 2017). R es uno de los lenguajes
más empleados en ciencias naturales en la actualidad (Cayuela y de la Cruz 2022). Esto se debe,
entre otros motivos, a su curva de aprendizaje sencilla en comparación con
otros lenguajes de programación, a la facilidad para hacer análisis
estadísticos y a la creación de gráficos visualmente llamativos con pocas
líneas de código, así como a la amplia comunidad que colabora en su desarrollo
(Giorgi et al. 2022).
R se estructura en paquetes, que son conjuntos de funciones
y de datos. R base cuenta con 15 paquetes por defecto (base, compiler,
datasets, grDevices, graphics, grid, methods,
parallel, splines, stats, stats4, tcltk, tools,
translations, y utils) (R Core Team 2023)
que permiten realizar trabajos de gestión de bases de datos, operaciones,
análisis de inferencia estadística y representación de gráficos, entre otros.
Sin embargo, la gramática, sintaxis y nomenclatura pueden variar entre estos
paquetes, lo que dificulta la migración del flujo de trabajo entre ellos (Çetinkaya-Rundel et al. 2022). Con el fin
de superar esta limitación, surge la filosofía de tidyverse, que busca
crear un entorno de trabajo en el que haya consistencia entre paquetes,
facilitando así la transición entre ellos durante las diferentes fases del
procesamiento de datos (Wickham et al. 2019; Çetinkaya-Rundel et al. 2022).
El objetivo de esta nota es introducir el conjunto de
paquetes del núcleo de tidyverse, abordando su origen, la filosofía que
respalda su creación y sus perspectivas futuras. A través de la comparación de
flujos de trabajo entre R base y tidyverse, se muestran diversas
formas de realizar una misma acción y a su vez, se aporta material para que los
usuarios interesados en profundizar en el mundo de tidyverse puedan
hacerlo de manera sencilla.
¿Qué es tidyverse?
Tidyverse es una colección de paquetes (meta-paquete)
de R (Wickham et al. 2019). El núcleo contiene 8
paquetes principales (readr, tibble, dplyr, tidyr, stringr, forcats,
ggplot2, y purrr), que comparten el mismo diseño, así como una
gramática y estructura de datos comunes. En el centro de la filosofía de tidyverse
se encuentra la noción de “datos ordenados” (“Tidy Data”). Wickham (2014) los define como “fáciles de manipular,
modelar y visualizar, y que tienen una estructura específica: cada variable es
una columna, cada observación es una fila y cada valor es una celda”. Además de
la estructura de los datos, tidyverse puede considerarse un dialecto del
lenguaje de programación R que discretiza funciones amplias de R base
(p. ej. merge) en diversas funciones específicas (p. ej. left_join, right_join)
(Staples 2023).
A continuación, se muestran cuatro principios básicos para
una introducción sencilla a este meta-paquete (https://design.tidyverse.org/unifying.html):
Centrado en humanos: tiene un diseño intuitivo que facilita su
uso y accesibilidad para el usuario, estando diseñado específicamente para
respaldar las actividades de un analista de datos humano.
Consistencia: todos los paquetes están diseñados para trabajar
con datos ordenados (“Tidy Data”), y las funciones de los paquetes tienen una
nomenclatura común y congruente. Esto permite que un usuario que aprende sobre
una función o paquete pueda aplicar ese conocimiento a otros, facilitando así
la tarea de recordar y deducir código de manera más sencilla.
Componiblidad: permite al usuario resolver problemas complejos
dividiéndolos en pequeñas partes, que pueden combinarse utilizando el operador “pipe”,
ya sea el del paquete magrittr (%>%) (Bache
y Wickham 2022) o el creado recientemente en R base (|>).
Inclusividad: no se trata sólo de la colección de paquetes, sino
también la comunidad de personas que los usan, basándose en la ayuda entre
usuarios y generando un ambiente inclusivo. Dos ejemplos destacados de este
último principio son la existencia de una guía de estilo para el flujo de
trabajo y escritura de código (https://style.tidyverse.org/index.html)
o el desarrollo de un método específico para solicitar ayuda a la comunidad y
obtener asesoramiento mediante ejemplos reproducibles (reprex) (Bryan et al. 2024).
Núcleo de tidyverse
En este apartado se presenta cada uno de los 8 paquetes del
núcleo de tidyverse con una breve descripción y código para ejemplificar
el flujo de trabajo que se puede seguir en cualquier procedimiento de análisis
de datos (Fig. 1).
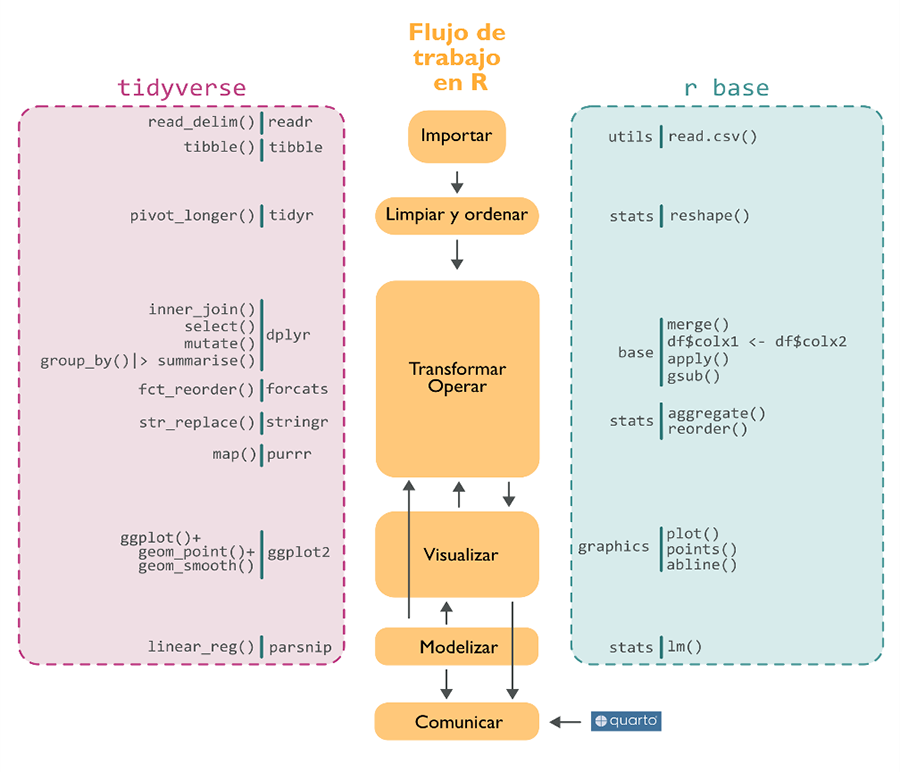
Figura 1. Esquema comparativo entre tidyverse y
R base donde se muestra el flujo de trabajo y funciones de ejemplo para cada
paso de análisis de datos desde su importación en R hasta la comunicación de
los resultados obtenidos.
Figure 1. Comparative diagram between tidyverse and base R showing the workflow and example functions for each step of data analysis from dataset import into R to the final step of reporting results.
Para empezar, se requiere cargar tidyverse y el conjunto de
datos de ejemplo, palmerpenguins (Horst et al. 2020).
library(palmerpenguins)
library(tidyverse)
Para garantizar la reproducibilidad
del código mostrando un flujo de trabajo desde el inicio, importando los datos
desde un fichero externo, es necesario exportar los datos de ejemplo a archivo
de valores separado por comas (.csv) previamente.
# tidyverse
write_csv(penguins, "penguins1.csv")
# R base
write.csv(penguins, "penguins2.csv")
- readr:
proporciona una forma rápida y sencilla de leer y guardar datos rectangulares,
como archivos de valores separados por comas (.csv) o archivos de texto delimitados
por tabuladores (.txt). https://readr.tidyverse.org/
# leer
archivos
penguins_tb <- read_csv("penguins1.csv")
# R base
penguins_df <-
read.csv("penguins2.csv")
- tibble:
introduce una nueva estructura de datos denominada “tibble(tbl_df)” que es una
versión moderna del “data.frame”, manteniendo las características de los
“data.frame” que han resultado útiles a lo largo del tiempo y mejorando las que
han resultado ser más problemáticas. Por ejemplo, no cambia el nombre o tipo de
variable y no realiza coincidencias parciales. Además, con la función
“print()”, un “tibble” muestra más información acerca de las variables que un
“data.frame”. https://tibble.tidyverse.org/
# lo lee como un
tibble
head(penguins_tb, n = 3)
# A tibble: 6 x 9
...1 species island
bill_length_mm bill_depth_mm flipper_length_mm
<dbl>
<chr> <chr> <dbl>
<dbl> <dbl>
1 1 Adelie
Torgersen 39.1 18.7 181
2 2 Adelie
Torgersen 39.5 17.4 186
3 3 Adelie
Torgersen 40.3 18 195
# R base lo lee como un data frame
head(penguins_df, n = 3)
X species
island bill_length_mm bill_depth_mm flipper_length_mm
1 1 Adelie
Torgersen 39.1 18.7 181
2 2 Adelie
Torgersen 39.5 17.4 186
3 3 Adelie
Torgersen 40.3 18.0 195
- dplyr:
agrupa las principales funciones necesarias para la manipulación de datos,
tales como filtrar datos, resumir, añadir o seleccionar nuevas variables, etc.,
en un conjunto de funciones con nombres intuitivos. Se enfoca en la simplicidad
y el rendimiento, permitiendo realizar operaciones complejas con una sintaxis
clara y concisa. https://dplyr.tidyverse.org/
# calcular la media de la
longitud y profundidad del pico por isla, especie y año
penguins_tb_mean
<- penguins_tb |>
group_by(island,
species, year) |>
summarise(bill_length_mm_mean
= mean(bill_length_mm, na.rm = TRUE),
bill_depth_mm_mean = mean(bill_depth_mm, na.rm = TRUE),
.groups =
"drop")
# R base
penguins_df_mean <-
aggregate(cbind(bill_length_mm, bill_depth_mm) ~ island + species + year,
data = penguins_tb,
FUN = function(x) mean(x, na.rm = TRUE))
colnames(penguins_df_mean)[4:5]
<- c("bill_length_mm_mean", "bill_depth_mm_mean")
- tidyr: está
diseñado para facilitar la organización y limpieza de datos, facilitando la
creación de datos ordenados para realizar procesos posteriores (análisis,
representación, etc.). Uno de sus objetivos principales es el intercambio entre
bases de datos en formato “ancho” (e.g., matriz) y formato “largo” (e.g., bases
de datos ordenadas). https://tidyr.tidyverse.org/
# organizar los
datos en formato largo en base a la longitud y profundidad del pico
penguins_tb_mean_long
<- penguins_tb_mean |>
pivot_longer(cols = c(bill_length_mm_mean, bill_depth_mm_mean),
names_to = "bill_variable",
values_to = "bill_value")
# R base
penguins_df_mean_long
<- reshape(
data =
penguins_df_mean,
varying =
list(c("bill_length_mm_mean", "bill_depth_mm_mean")),
v.names =
"bill_value",
times =
c("bill_length_mm_mean", "bill_depth_mm_mean"),
timevar =
"bill_variable",
direction =
"long"
)
rownames(penguins_df_mean_long)
<- NULL
penguins_df_mean_long$id
<- NULL
- stringr:
está diseñado para simplificar y hacer más eficiente la manipulación y el
procesamiento de cadenas de texto. Proporciona funciones con nomenclatura y
gramática muy intuitiva (p. ej., todas las funciones empiezan por “str_...()”)
para trabajar con cadenas de texto, como búsqueda, reemplazo, extracción y
modificación de patrones en textos. https://stringr.tidyverse.org/
# extraer las dos primeras
palabras de la variable que hemos creado uniendo la longitud y profundidad del
pico
penguins_tb_mean_long_str
<- penguins_tb_mean_long |>
mutate(bill_str =
word(bill_variable, start = 1L, end = 2L, sep = "_"))
# R base
penguins_df_mean_long$bill_str
<- sapply(strsplit(penguins_df_mean_long$bill_variable, "_"),
function(x)
paste(x[1:2], collapse = "_"))
penguins_df_mean_long_str <-
penguins_df_mean_long
- forcats:
permite la manipulación de variables de tipo factor (variables categóricas que
pueden tomar un número limitado de valores distintos, conocidos como niveles).
Es especialmente útil cuando se quiere ordenar los niveles del factor, por
ejemplo, para representar gráficamente resultados en un determinado orden que
facilita la visualización. https://forcats.tidyverse.org/
# reordenar los
niveles del factor especies manualmente
penguins_tb_mean_long_str_for
<- penguins_tb_mean_long_str |>
mutate(species = fct_relevel(species, c("Chinstrap", "Adelie",
"Gentoo")))
# fct_relevel() no altera
directamente el orden de los niveles en la tabla,
# sino* que los reordena
internamente para determinados procesos, como por ejemplo, una
# mejor visualizacion en los
graficos
# R base
penguins_df_mean_long_str$species
<- factor(penguins_df_mean_long_str$species,
levels = c("Chinstrap", "Adelie", "Gentoo"))
penguins_df_mean_long_str_for
<- penguins_df_mean_long_str
- ggplot2:
permite representar datos gráficamente. Su filosofía se basa en el
funcionamiento por capas. Así, una vez definido el origen de los datos y las
variables, con el operador “+” se añaden el tipo de gráfico (puntos, líneas,
boxplots, etc.) y otras características adicionales (títulos, fondo, ejes
etc.). https://ggplot2.tidyverse.org/
# generar una
grafica mostrando la longitud y profundidad del pico de cada especie
ggplot(penguins_tb_mean_long_str_for,
aes(x = bill_str, y = bill_value, color = species)) +
scale_color_manual(values = c("red", "green",
"blue")) +
geom_boxplot() +
labs(x = "variable", y = "mm")
# R base
boxplot(bill_value
~ species * bill_str,
data =
penguins_df_mean_long_str_for,
xlab =
"species & variable", ylab = "mm", col = c("red",
"green", "blue"))
- purrr:
proporciona un conjunto completo de herramientas para trabajar con funciones y
vectores, enfocado desde la programación funcional. Sus funciones más
representativas son las del conjunto “map()”, que se presentan como alternativa
a la programación imperativa, la cual incluye herramientas como los bucles
“for” y “while”. Mediante el enfoque de programación funcional se genera código
más conciso, modular, y fácil de reutilizar y de leer por el usuario. https://purrr.tidyverse.org/
# generar una
grafica para cada especie
plot_species_tidy
<- function(species_name) {
penguin_species_plot <- penguins_tb_mean_long_str_for
|>
filter(species == species_name) |>
ggplot(aes(x = bill_str, y = bill_value)) +
geom_boxplot() +
labs(x =
"variable", y = "mm") +
ggtitle(species_name)
return(penguin_species_plot)
}
map(.x =
levels(penguins_tb_mean_long_str_for$species),
.f = plot_species_tidy)
# R base
plot_species_base
<- function(species_name) {
species_data <- subset(penguins_df_mean_long_str_for, species ==
species_name)
penguin_species_plot <- boxplot(bill_value ~ bill_str, data = species_data,
xlab = "variable", ylab = "mm",
main = species_name)
return(penguin_species_plot)
}
lapply(X =
levels(penguins_df_mean_long_str_for$species),
FUN = plot_species_base)
Por último, para finalizar este
apartado, se presenta un ejemplo donde se puede ver la funcionalidad que
presenta tidyverse para ejecutar varias acciones en una misma línea
usando el operador “pipe”.
# grafica de tendencia media
anual de masa corporal para cada especie
plot_mass_island
<- read_csv("penguins1.csv") |>
group_by(year,
species) |>
summarise(body_mass_g = mean(body_mass_g, na.rm = TRUE)) |>
ggplot(aes(x =
year, y = body_mass_g, col = species)) +
geom_line() +
scale_x_continuous(breaks = c(2007, 2008, 2009)) +
labs(x = "year", y
= "mean body mass (g)")
plot_mass_island
# R base
penguins_df <-
read.csv("penguins2.csv")
aggregated_data
<- aggregate(body_mass_g ~ year + species, data = penguins,
FUN = function(x) mean(x, na.rm = TRUE))
par(mar = c(5, 4, 4, 8), xpd =
TRUE)
plot(aggregated_data$year,
aggregated_data$body_mass_g, type = "n", xlab = "year",
ylab = "mean body mass (g)", xaxt = "n")
species_data_list <-
split(aggregated_data, aggregated_data$species)
plot_species_line
<- function(data, color) {
lines(data$year,
data$body_mass_g, col = color, type = "o")
}
species_colors <-
c("Adelie" = "red", "Chinstrap" =
"green", "Gentoo" = "blue")
lapply(names(species_data_list),
function(species) {
plot_species_line(species_data_list[[species]], species_colors[[species]])
})
axis(1, at = c(2007, 2008,
2009))
legend("topright",
inset = c(-0.3, 0.3),
legend =
names(species_colors), col = species_colors, lty = 1, title =
"Species")
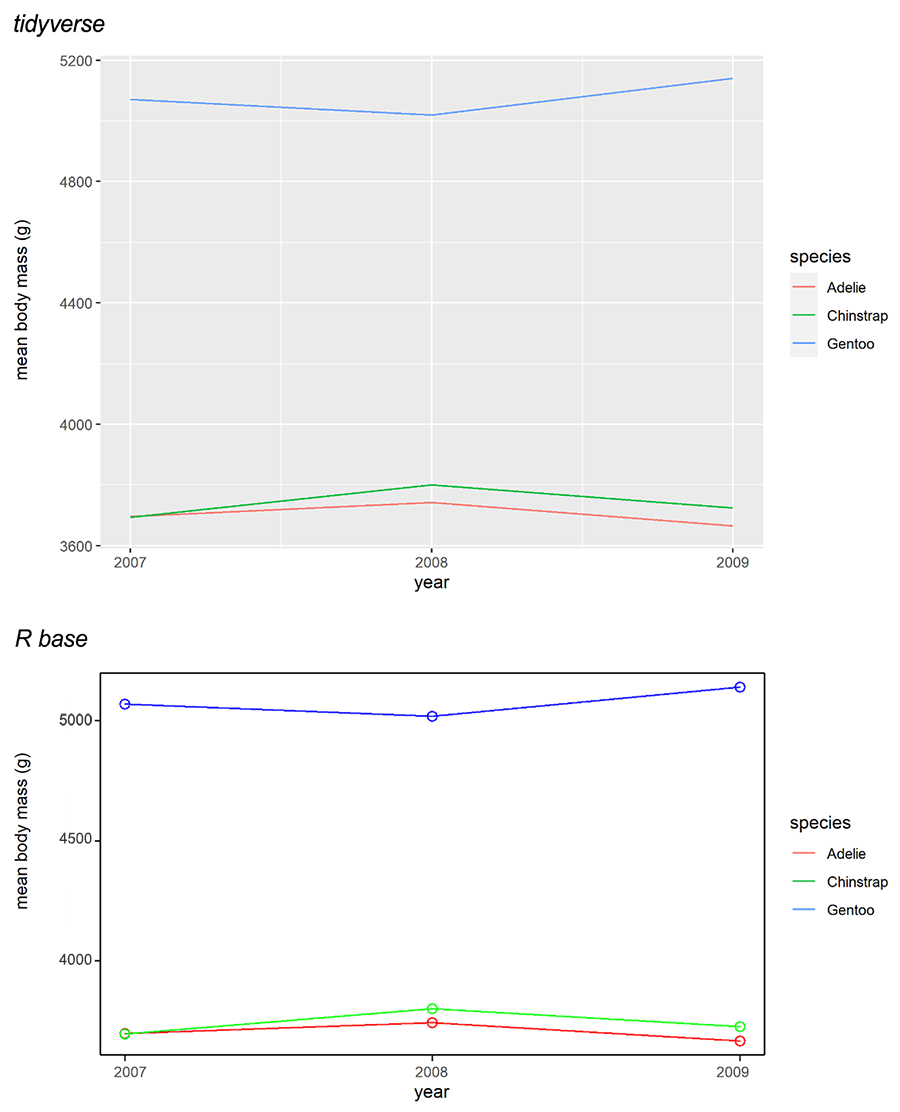
Figura 2. Gráficos obtenidos mediante tidyverse
(arriba) y R base (abajo)
Figure 2. Plots obtained from tidyverse (top panel) and base R (bottom
panel).
Actualidad y futuro de tidyverse
Como sucede con cualquier lenguaje relativamente nuevo, R
está experimentando un rápido proceso de evolución en el que tidyverse
va ganando presencia. En esta nota se han descrito las funcionalidades que lo
convierten en un meta-paquete con gran potencial para el procesado de datos
desde un nivel básico, pero también puede presentar algunas limitaciones. Por
ejemplo, al tratarse de varios paquetes interconectados, actualizar alguno de
ellos a veces puede causar problemas de compatibilidad con otros paquetes o con
el propio código escrito anteriormente. Las funciones de tidyverse se
actualizan a un ritmo más rápido que las de R base, lo que puede
requerir actualizar el código con mayor frecuencia y estar al tanto de las
nuevas actualizaciones como usuarios.
El auge de tidyverse ha llevado a una división de
opiniones entre los usuarios de R acerca de su uso. Por un lado, hay usuarios
que comenzaron su formación con R base y el uso de tidyverse les
resulta menos intuitivo. Por otro lado, los
usuarios a favor del uso de tidyverse argumentan que, para obtener un
mismo resultado, la gramática empleada es más fácil de entender y asimilar,
especialmente para principiantes. Aunque el auge de tidyverse podría
implicar una diferenciación que rompa la continuidad, consistencia y
aplicabilidad entre R base y tidyverse, en última instancia son
los usuarios quienes van moldeando la naturaleza de los lenguajes y
determinarán su futuro (Staples
2023). Nuestra intención no es entrar en el marco de debate tidyverse
vs. R base, pues no se pretende abogar por el uso de uno frente a otro,
sino presentar el meta-paquete de tidyverse y adjuntar código
comparativo con R base para mostrar y facilitar el aprendizaje a
aquellas personas que estén interesadas. Existen numerosas posibilidades para
ejecutar una misma acción, por lo que el código que se muestra es una opción de
las múltiples formas posibles de llegar al mismo resultado. En última
instancia, conocer la estructura de ambos enfoques nos permitirá alternar entre
las opciones más eficientes para cada paso de nuestros análisis.
Contribución de los autores
Olivia Lorente-Casalini: conceptualización, validación,
redacción – borrador original y redacción – revisión y edición. Marina
Rodes-Blanco: conceptualización, validación, redacción – borrador original y
redacción – revisión y edición. Sofía Aguirre-Iglesias: conceptualización y
redacción – revisión y edición. Jorge A. Martín-Ávila: conceptualización y
redacción – revisión y edición. Darío San-Segundo Molina: conceptualización,
visualización y redacción – revisión y edición. Julián Tijerín-Triviño: conceptualización
y redacción – revisión y edición. Julen Astigarraga: conceptualización,
supervisión y redacción – revisión y edición.
Agradecimientos
OLC está financiada por el Programa de Contratación de
Personal Investigador Predoctoral en Formación de la Comunidad de Madrid (PIPF
2022-PEJ); MRB por los proyectos VERDAT (Organismo Autónomo de Parques
Nacionales, MITECO, Ref. 2794/2021) y REMOTE (Ministerio de Ciencia e
Innovación, PID2021-123675OB-C42); SAI por el proyecto IBERIAN FUTURE WINES de
la Comunidad de Madrid en el marco del Convenio Plurianual con la Universidad
de Alcalá en la Línea de actuación “Programa de Fondos de Investigación para
las Ayudas Beatriz Galindo” (CM/BG/2021-003); JAMA está financiado por un
contrato FPI (Ministerio de Ciencia e Innovación, PRE2020-093652); DSSM por el
Programa de Formación del Profesorado Universitario del Ministerio de
Universidades (FPU20/05528); JTT está financiado por el proyecto VERDAT
(Organismo Autónomo de Parques Nacionales, MITECO, Ref. 2794/2021) y por el
Programa de Estímulo a la Excelencia para el Profesorado Universitario
Permanente (EPU-INV/2020/010) y JA por el proyecto CLIMB-FOREST Horizon Europe
(nro. 101059888) financiado por la Unión Europea. Queremos agradecer al grupo
de Ecoinformática de la AEET, especialmente a Verónica Cruz-Alonso, Julia G. de
Aledo y Antonio Canepa, por sus sugerencias para mejorar esta nota. Esta nota
se ha revisado siguiendo un proceso colaborativo y público disponible en: https://github.com/ecoinfAEET/Notas_Ecosistemas/issues/56
Esta nota es el resultado del
grupo de trabajo creado por estudiantes de doctorado de la Unidad Docente de
Ecología de la Universidad de Alcalá para profundizar en el uso de herramientas
relacionadas con el análisis de datos. Para profundizar más en la comparación tidyverse
vs. R base, se puede consultar el repositorio creado para esta
nota ecoinformática. (https://github.com/OliviaLorente-Casalini/nota_tidyverse)
Referencias
Bache, S.,
Wickham, H. 2022. _magrittr: A Forward-Pipe
Operator for R_. R package version 2.0.3. https://CRAN.R-project.org/package=magrittr
Bezanson, J., Edelman, A., Karpinski, S., Shah, V.B. 2017. Julia: A Fresh
Approach to Numerical Computing. SIAM Review 59(1): 65–98. https://doi.org/10.1137/141000671
Bryan, J., Hester, J., Robinson, D., Wickham, H., Dervieux, C. 2024. _reprex:
Prepare Reproducible Example Code via the Clipboard_. R package version
2.1.0. https://CRAN.R-project.org/package=reprex.
Cayuela, L., de la Cruz,
M. 2022. Análisis de datos ecológicos en R. Ediciones Mundi-Prensa. Madrid,
España.
Çetinkaya-Rundel, M., Hardin, J., Baumer, B.S., McNamara, A., Horton, N.J., Rundel,
C. 2022. An educator”s perspective of the tidyverse. Technology
Innovations in Statistics Education 14(1). https://doi.org/10.5070/T514154352
Ellwood, E.R., Sessa, J.A., Abraham, J.K., Budden, A.E., Douglas, N.,
Guralnick, R., Krimmel, E., et al. 2020. Biodiversity Science and the
Twenty-First Century Workforce. BioScience 70(2): 119–121. https://doi.org/10.1093/biosci/biz147
Fanelli, D. 2018. Is science really facing a reproducibility crisis, and do
we need it to? Proceedings of the National Academy of Sciences 115(11):
2628–2631. https://doi.org/10.1073/pnas.1708272114
Farley, S.S., Dawson, A., Goring, S.J., Williams, J.W. 2018. Situating
Ecology as a Big-Data Science: Current Advances, Challenges, and Solutions. BioScience
68(8): 563–576. https://doi.org/10.1093/biosci/biy068
Giorgi, F.M., Ceraolo, C., Mercatelli, D. 2022. The R Language: An Engine
for Bioinformatics and Data Science. Life 12(5), 648. https://doi.org/10.3390/life12050648
Hampton, S.E., Strasser, C.A., Tewksbury, J.J., Gram, W.K., Budden, A.E.,
Batcheller, A.L., Duke, C.S., et al. 2013. Big data and the future of ecology. Frontiers
in Ecology and the Environment 11(3): 156–162. https://doi.org/10.1890/120103
Horst, A., Hill, A., Gorman, K. 2020. palmerpenguins: Palmer
Archipelago (Antarctica) penguin data. R package version 0.1.0. https://allisonhorst.github.io/palmerpenguins
/ https://doi.org/10.5281/zenodo.3960218
King, G. 2011. Ensuring the Data-Rich Future of the Social Sciences. Science
331(6018): 719–721. https://doi.org/10.1126/science.1197872
Michener,
W.K., Jones, M.B. 2012. Ecoinformatics: supporting
ecology as a data-intensive science. Trends in Ecology & Evolution
27(2): 85–93. https://doi.org/10.1016/j.tree.2011.11.016
R Core Team 2023. _R: A Language and Environment for Statistical Computing_.
R Foundation for Statistical Computing, Vienna, Austria. https://www.R-project.org/.
Staples,
T.L. 2023. Expansion and evolution of the R programming language. Royal
Society Open Science 10(4): https://doi.org/10.1098/rsos.221550
Van Rossum,
G., Drake, F.L. 1995. Python
reference manual. Centrum voor Wiskunde en
Informatica Amsterdam. The Netherlands.
Wickham, H. 2014. Tidy Data. Journal of Statistical Software 59(10):
1-23. https://doi.org/10.18637/jss.v059.i10
Wickham, H., Averick, M., Bryan, J., Chang, W., D´Agostino Mcgowan, L.,
François, R., Grolemund, G., et al. 2019. Welcome to the Tidyverse. Journal
of Open Source Software 4(43): 1686. https://doi.org/10.21105/JOSS.01686
Wickham, H., Çetinkaya-Rundel, M., Grolemund, G. 2023. R for data
science: Import, tidy, transform, visualize and model data (2nd ed.). O”Reilly
Media, Inc. Sebastopol, CA, USA.
![]() , Marina Rodes-Blanco1,2,*
, Marina Rodes-Blanco1,2,* ![]() , Sofía
Aguirre-Iglesias3
, Sofía
Aguirre-Iglesias3 ![]() , Jorge A. Martín-Ávila1
, Jorge A. Martín-Ávila1 ![]() ,
Darío San-Segundo Molina3
,
Darío San-Segundo Molina3 ![]() , Julián Tijerín-Triviño1
, Julián Tijerín-Triviño1 ![]() , Julen Astigarraga1
, Julen Astigarraga1 ![]()

