Introducción
Los géneros de la familia Pinaceae son árboles siempre
verdes que constituyen un componente dominante de los bosques de alta montaña
en el hemisferio Norte (Fig. 1; Farjon y Filer 2013). En Norteamérica se
distribuyen un total de seis géneros (Abies Miller, Larix Miller,
Picea A. Dietr., Pinus L., Pseudotsuga Carrière y Tsuga
(Endlicher) Carrière)); sin embargo, en esta revisión se consideraron sólo los
cuatro géneros con una distribución latitudinal más amplia, estos son: Abies,
Picea, Pinus y Pseudotsuga. Los estudios en Pinaceae
indican que sus especies poseen altos niveles de diversidad genética (DG) (Ivetić y Devetaković 2017). La evolución de
una especie o de las poblaciones depende enormemente del nivel de polimorfismo
genético y de los patrones de distribución de la DG (Haselhorst et al. 2019), cuantificables
dentro y entre poblaciones (Ivetić y
Devetaković 2017).
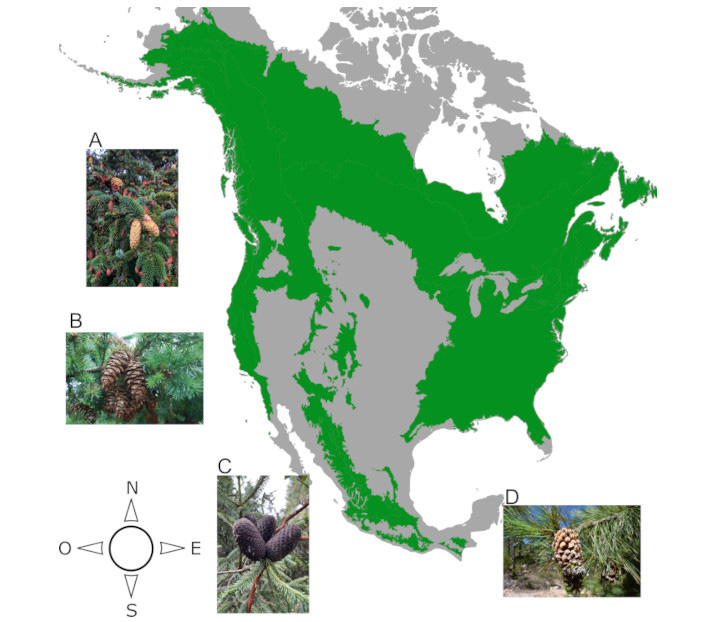
Figura 1. Distribución de los bosques de la
familia Pinaceae en Norteamérica. Nombre y localización de las fotos de algunas
de las especies de los cuatro géneros estudiados: A) Picea sitchensis
(Oregon); B) Pseudotsuga menziessi (Oregon); C) Abies
religiosa (Hidalgo); D) Pinus strobiformis (Chihuahua). Fotografías por David S. Gernandt.
Figure 1. Distribution of Pinaceae forests in North America. Name and photos location of some of the
species of the four genera studied: A) Picea sitchensis (Oregon); B) Pseudotsuga menziessi (Oregon); C) Abies religiosa (Hidalgo); D) Pinus strobiformis (Chihuahua). Pictures by David S. Gernandt.
La DG puede ser influenciada también por la hibridación, ya
que es un proceso que puede introducir nuevo material genético a las
poblaciones y resultar en especiación (Mallet 2008).
Este proceso se ha detectado en especies estrechamente relacionadas y con
mecanismos de aislamiento reproductivo débiles (Delgado
et al. 2007; Soltis y Soltis 2009). Las
estimaciones de DG en Pinaceae se han realizado en base a diversos marcadores
moleculares. Los marcadores citoplasmáticos como la mitocondria y el
cloroplasto son de herencia uniparental. En este caso, la mitocondria se hereda
vía materna y el cloroplasto vía paterna, mientras que los marcadores nucleares
son de herencia biparental y están sujetos a la recombinación (Tsutsui et al. 2009). En la mitocondria se han
detectado bajas tasas de sustitución molecular y secuencias repetitivas
complejas dispersas en distintas regiones y estructuras quiméricas que
posiblemente fueron generadas por múltiples recombinaciones intergenómicas (Smith 2016). El cloroplasto también evoluciona
lentamente y exhibe baja variación a nivel de especie y poco o nada de
recombinación (Olmstead y Palmer 1994). En el genoma del
cloroplasto hay regiones no codificantes que contienen marcadores variables,
como los microsatélites (SSRs; por sus siglas en inglés), los cuales son
ampliamente usados por su alto polimorfismo (Vendramin
et al. 1996). Por lo tanto, el uso de los distintos marcadores moleculares
puede reflejar la DG de diferentes regiones del genoma (Xu
et al. 2008), de gran importancia para complementar la historia evolutiva
de las especies.
Consideramos que para lograr un mejor entendimiento de los
procesos microevolutivos asociados a los niveles de DG presentes en este grupo
de árboles, es importante contar con un análisis comparativo de las
investigaciones que se han realizado con el uso de diferentes marcadores
moleculares. En este trabajo de revisión bibliográfica se describen los niveles
de DG con distintos marcadores moleculares para cuatro géneros de Norteamérica
(Abies, Picea, Pinus y Pseudotsuga), así como los
procesos microevolutivos que explican esta DG a la luz de los conocimientos
actuales. Los objetivos de este trabajo fueron: 1) realizar una síntesis sobre
los trabajos realizados de la diversidad genética en cuatro géneros de la
familia Pinaceae en Norteamérica, con el fin de documentar los patrones y
procesos microevolutivos que expliquen la diversidad genética, 2) utilizar la
información de DG y de la estructura genética de las especies para determinar
si estas se encuentran correlacionadas significativamente, con lo cual esperaríamos
que en especies con mayor estructuración genética se encuentren menores valores
de DG y, 3) evaluar a través de los modelos lineales generalizados (GLM; por
sus siglas en inglés) si las variables especie y marcador molecular explican la
DG en las especies.
Patrones de
diversidad genética en el género Abies
En Norteamérica se reconocen 18 especies, de las cuales al
menos en 12 especies se han realizado estudios de genética de poblaciones,
representando el 66.6% del género Abies (Flora of North
America, [utilizado el 10 de Julio, 2024]; Gernandt y Pérez de la Rosa 2014).
Diversas investigaciones sugieren que las especies del género Abies
contienen mayor DG dentro de las poblaciones y una menor variación
inter-poblacional. Por ejemplo, para Abies balsamea (L.) Mill, a través
de un análisis de isoenzimas, se obtuvieron 96.3% y 3.7% de DG dentro y entre
poblaciones respectivamente (Shea y Furnier 2002).
Similarmente en A. procera Rehder, con polimorfismos de nucleótido único
(SNPs; por sus siglas en inglés) del genoma nuclear, 96% de DG dentro de las
poblaciones y 4% entre las poblaciones (Hansen et al.
2020).
En el género Abies los niveles de DG reportados
varían dependiendo del marcador utilizado (Anexo, Tabla
A1). Sin embargo, comparaciones con los mismos marcadores nos permiten
identificar aquellas especies con mayor diversidad intrapoblacional. Por
ejemplo, en un complejo de especies de abetos mexicanos, la DG obtenida con
isoenzimas en la especie Abies flinckii Rushforth (Heterocigosis
esperada, He = 0.113), cuenta con la mayor DG, en comparación con, A.
guatemalensis Rehder (He = 0.069), A. hickelii Flous &
Gaussen (He = 0.100) y A. religiosa A.E. Murray (He =
0.108) (Aguirre-Planter et al. 2000).
Mientras que las especies de Estados Unidos, mostraron valores similares (A.
grandis (Douglas ex D.Don) Lindl., isoenzimas, He = 0.13, Konnert y Ruetz 1997; A. balsamea,
isoenzimas, He = 0.018, Shea y Furnier 2002).
De los abetos norteamericanos, resalta A. religiosa
como la especie más estudiada, debido a su fácil reconocimiento en el campo y
su amplia distribución geográfica. La DG obtenida con marcadores de
mitocondria, secuencias de cloroplasto y núcleo, y microsatélites de
cloroplasto, fue variable, en rangos de 0.29 a 0.86 (Arzate-Fernández et al. 2016; Cruz-Nicolás et al. 2019). Esta alta diversidad
se ha encontrado con información de SSRs en A. lasiocarpa (Hook.) Nutt.,
He = 0.642 y sus variedades, A. lasiocarpa var. bifolia
(A. Murray) Silba, He = 0.683 y A. lasiocarpa var. arizonica (Merriam)
Lemmon, He = 0.697 (Ritland et al. 2022).
El porcentaje de polimorfismo (P) con marcadores
isoenzimas varió del 13 al 43%, lo cual es menor a lo reportado en otras
especies de Pinaceae (Ledig et al. 1999; Viveros-Viveros et al. 2010). No obstante,
las estimas más bajas de porcentaje de polimorfismo corresponden a especies con
una distribución geográfica reducida (por ejemplo, A. bracteata (D. Don)
Poiteau (P = 13.3%, Ledig et al. 2006), A.
procera (P = 40%, Yeh y Hu 2005), A. guatemalensis (P =
20%) o A. hickelii (P = 28.2%, Aguirre-Planter
et al. 2000). Contrario a A. religiosa, una especie de amplia
distribución geográfica, donde se encuentran altos niveles de polimorfismo con
distintos marcadores moleculares (isoenzimas, P = 90%; secuencias de
cloroplasto, P = 95%, de mitocondria, P = 100% y nucleares, P =
100%; Arzate-Fernández et al. 2016).
Los estudios con isoenzimas y secuencias del genoma
mitocondrial sugieren que los abetos mexicanos (A. flinckii, A.
guatemalensis, A. hickelii y A. religiosa) han pasado por
cuellos de botella (Jaramillo-Correa et al.
2008; Arzate-Fernández et al. 2016). También, se han reportado resultados
similares en abetos de Estados Unidos, por ejemplo, en A. lasiocarpa (He
= 0.111), A. balsamea (He = 0.018) y A. procera (He
= 0.22) (Ettl y Peterson 2001; Hansen et al. 2020), donde los bajos niveles de DG
pueden ser el resultado de una contracción y subsecuente expansión del tamaño
efectivo poblacional (Ne) a partir de un refugio glacial durante el
Último Máximo Glacial (LGM, por sus siglas en inglés).
En este género se han reportado casos de poblaciones
híbridas con mayor DG con respecto a sus parentales. Por ejemplo, en zonas
alopátricas de A. balsamea, la DG del ADN mitocondrial (ADNmt) dentro
de las poblaciones es nula, (Hs = 0), al igual que en A.
lasiocarpa (ADNmt = zona alopátrica; Hs = 0), mientras
que en la zona de contacto de ambas especies la DG es mayor (Hs =
0.25) (Cinget et al. 2015). Los niveles de DG detectados en mitocondria son
contrastantes con los obtenidos analizando el cloroplasto (A. balsamea,
ADNcp = zona alopátrica (Hs = 0.74); A. lasiocarpa,
ADNcp = zona alopátrica (Hs = 0.97); zona de contacto (Hs
= 0.88) (Cinget et al. 2015). Estas
tendencias han sido similares a otras investigaciones que abordan esta temática
con otras especies de pináceas (por ejemplo, Marquardt
et al. 2007; Godbout et al. 2012; Cruz-Nicolás et al. 2019).
Patrones de diversidad genética en el género Picea
Este género alberga en Norteamérica 10 especies, de las
cuales en 7 especies se han documentado estudios de genética de poblaciones,
representando el 70% del total de especies incluidas en esta sección (Flora of North America, [utilizado el 10 de Julio, 2024]; Gernandt y Pérez de la Rosa 2014). En
el género Picea se observan patrones de DG similares a otras pináceas.
Las especies de este género contienen la mayor DG dentro de sus poblaciones y
poca diferenciación entre poblaciones. Este patrón se observa incluso en
especies con poblaciones fragmentadas, como P. breweriana S. Watson, con
base en isoenzimas, con un 84.8% dentro y 15.2% de variación entre poblaciones
(Ledig et al. 2006), P. chihuahuana Martínez,
con secuencias de cloroplasto y mitocondria, con 70% dentro y 30% entre las
poblaciones (Jaramillo-Correa et al. 2006),
P. martinezii Patterson, con base en isoenzimas, con 90% dentro y 10%
entre las poblaciones (Ledig et al. 2002).
En especies con menor fragmentación la variación entre poblaciones puede ser
incluso menor, como es el caso de P. sitchensis (Bongard) Carrière, con
SNPs, 97.1% dentro y 2.9% entre poblaciones (Byrne et
al. 2022). En la Tabla A2 del anexo se
encuentran reportados las cuantificaciones de la diversidad genética de las
especies del género Picea revisadas en esta sección.
La cuantificación de la DG de marcadores mitocondriales
difiere con otros marcadores, porque la variación mitocondrial refleja el flujo
mediado por el movimiento de semillas, mientras que el cloroplasto está mediado
por el movimiento a larga distancia del polen. Por ejemplo, Jaramillo-Correa et al. (2006) encontraron
ausencia de variación mitocondrial en P. chihuahuana (He = 0),
mientras que con marcadores de cloroplasto los niveles de DG fueron diferentes
para la distribución norte (He = 0.548), centro (He = 0.300) y
sur (He = 0.167). Las especies P. martinezii (isoenzimas, He =
0.08; Ledig et al. 2002) y P. mexicana
Martínez (SNPs, He = 0.2; Mendoza-Maya et
al. 2015) muestran valores de DG comparables con P. chihuahuana. La
baja DG detectada con isoenzimas sugiere que las especies tienen una
deficiencia de heterocigotos. Este patrón es observado también en especies de
Estados Unidos, con isoenzimas en P. glauca (Moench) Voss (Tremblay y Simon 1989) y microsatélites
nucleares (SSRn; por sus siglas en inglés) (Heterocigosis observada, Ho =
0.49 y He = 0.637) (Fageria y Rajora 2013).
Las posibles causas a esta deficiencia de heterocigotos han sido varias: 1) la
selección en contra de los heterocigotos, 2) adaptación a nivel local de las
poblaciones, 3) endogamia y, 4) el efecto Wahlund (Ledig
et al. 2002; Rajora et al. 2005).
El porcentaje del polimorfismo obtenido con isoenzimas en
poblaciones de distribución geográfica reducida osciló entre 35.2% (P.
breweriana, Ledig et al. 2005) y 44.2% (P.
mexicana, Ledig et al. 2002), los cuales
fueron menores en especies de píceas con mayor distribución geográfica como P.
mariana (Mill.) Britton, Sterns & Poggenb. (P = 66.8%; Rajora y Pluhar
2003) o P. chihuahuana con el uso
de polimorfismos en la longitud de fragmentos amplificados (AFLPs; por sus
siglas en inglés) (P = 80%) que presentan poblaciones fragmentadas (Quiñones-Pérez et al. 2017). El bajo
polimorfismo encontrado se puede atribuir a procesos como contracciones
poblacionales causadas por cambios ambientales, o debido al incremento de la
endogamia, sobre todo en especies con distribución fragmentada.
Los análisis
filogeográficos sugieren que las poblaciones de P. mexicana expandieron
su distribución durante períodos fríos. No obstante, con la entrada del clima
cálido posterior al último máximo glacial (LGM) las poblaciones tuvieron un
declive y se fragmentaron (Ledig et al. 2002). De hecho, se encontró una baja DG (He = 0.125) en ellas, lo
cual fue asociado a eventos de cuellos de botella. La alta estructura genética
y la baja DG a consecuencia de la presencia de cuellos de botella, ha sido
reportada para otras píceas como P. breweriana (Ledig et al. 2005), P. rubens Sarg., P. engelmannii Engelm.
y P. pungens Engelm. con isoenzimas (Ledig et al. 2006) o con secuencias de cloroplasto en P. chihuahuana (Jaramillo-Correa
et al. 2006). Por otra parte, en
el género Picea también se han documentado casos de hibridación. En
estudios de P. glauca y P. engelmannii, se encontró una amplia
zona híbrida, con niveles moderados de DG formada por la introgresión
asimétrica y barreras reproductivas débiles entre las especies parentales
(isoenzimas, He = 0.184, Rajora y Dancik 2000; He = 0.255, Ledig et al. 2006; SSRn, He = 0.53; SNPs, He = 0.25; De la Torre et al.
2015). Una tendencia más clara la
presentaron las poblaciones híbridas de Picea sitchensis x P. glauca
estudiadas con SSRn, que mostraron niveles de DG más altos (He = 0.575),
con respecto a las poblaciones puras de P. sitchensis (He =
0.505) y P. glauca (He = 0.567) (Hamilton y
Aitken 2013), aunque la
cuantificación de la DG del grupo híbrido es comparable con P. glauca,
esto podría sugerir introgresión mayor de esta especie en la zona híbrida.
Patrones de diversidad genética en el género Pinus
El género Pinus es el más rico en la diversidad de
especies con respecto a los otros tres géneros que abordamos en esta revisión.
México representa uno de sus centros secundarios de diversificación al albergar
49 especies, más las 33 especies que se distribuyen en Estados Unidos de
América y Canadá, enlistadas en Flora of North
America [utilizado
el 10 de Julio, 2024]. No obstante, el número
reconocido para las especies de pinos en México podría incrementarse si
consideramos las variedades que reportan (Gernandt
y Pérez de la Rosa 2014). En esta sección se han documentado 29 estudios de
genética de poblaciones, lo que representa el 35.36% de las 82 especies
reportadas para Norte América, aunque este porcentaje puede ser mayor si se
consideran los estudios de las variedades que incluyen algunas especies. Los
estudios de DG realizados con diferentes tipos de marcadores moleculares en
este género sugieren también un patrón de mayor DG dentro que entre las
poblaciones, lo que puede ser consecuencia de los sistemas de apareamiento
cruzado y el modo de dispersión del polen a través del viento. Por ejemplo, con
la amplificación aleatoria de ADN polimórfico (RAPDs; por sus siglas en inglés)
Pinus cembroides Gordon y P. chiapensis (Martínez) Andresen,
presentaron niveles de DG de 85% y 77,4% dentro de las poblaciones y 15% y
22.6% entre poblaciones, respectivamente (Newton et
al. 2002; Aguirre-Limón et al. 2017),
en P. longaeva D.K. Bailey y P. rzedowskii (Madrigal &
Caballero) (con isoenzimas, de 98.9% y 82.5% dentro de las poblaciones y 0.011
y 17.5% entre poblaciones (Delgado et al. 1999; Lee et al. 2002) o con SSRn, 91% dentro y 9% entre
poblaciones de P. albicaulis Engelm (Lea et al.
2018). La Tabla A3 del anexo muestra
las diferentes investigaciones que han cuantificado la diversidad genética para
el género Pinus.
Pinus es el género con más especies en comparación
con los géneros Abies, Picea y Pseudotsuga y cuya
distribución geográfica puede ser amplia, restringida y/o fragmentada. Aunque
se espera que los niveles de DG sean bajos en las poblaciones fragmentadas (Niebling y Conkle 1990), especies mexicanas
con distribución fragmentada, tienen altos niveles de DG. Tal es el caso de P.
rzedowski, que, con el uso de isoenzimas, presentó una DG de He =
0.219 (Delgado et al. 1999), P. cembroides,
con las Inter Secuencias Simples Repetidas (ISSR; por sus siglas en inglés) con
He = 0.22 (Fuentes-Amaro et al. 2019). Así como en especies
de Estados Unidos, como P. balfouriana Balf., con He = 0.219,
determinada con isoenzimas (Hamrick et al. 1994),
P. longaeva, He = 0.33, con SSRn (Decker
2022). Estos valores son comparables con especies de pinos con amplia
distribución geográfica como P. monticola Douglas ex D. Don, estudiadas
con AFLPs (He = 0.235; Kim et al. 2010), P.
monticola, He = 0.269, obtenido con SNPs (Nadeau
et al. 2015) y P. engelmannii Carr., He = 0.23, con AFLPs (Morales-Nieto et al. 2021).
Los estudios revisados muestran diferentes niveles de
polimorfismo en los marcadores empleados. En P. maximartinezii Rzed.,
con isoenzimas los niveles del polimorfismo fueron de P = 30.3-33.3% (Ledig et al. 1997; Ledig et
al. 1999), en P. echinata Mill., de P = 87.2% (Raja 1997), P.
clausa (Chapm. ex Engelm) Sarg., P = 77.1%, P. clausa var. immuginata
D.B. Ward, P = 85.6% (Parker et al. 2001), en P. oocarpa Schiede ex Schltdl., P =
91.2% (Sáenz-Romero y Tapia-Olivares
2003) y, en P. strobus, P = 23.3% (Myers
et al. 2007). En P. chiapensis con RAPDs, el polimorfismo fue de P
= 30.3% (Newton et al. 2002) y en P.
longaeva, P = 63% (Lee et al. 2002). El
bajo polimorfismo encontrado en algunos estudios podría atribuirse a procesos
estocásticos como cuellos de botella o de efecto fundador, aunque esto tendría
que ser probado a la luz de los datos genómicos, que pueden ser obtenidos a
través de las nuevas plataformas de secuenciación masiva.
En contraparte, en otras especies como P. caribaea
var. hondurensis (Sénécl.) W.H. Barrett & Golfari (SSRn, Ho =
0.422 y 0.421, He = 0.465 y 0.488) (Delgado
et al. 2011; Rebolledo et al. 2018), se
han detectado declives en el Ne y como consecuencia, altos
niveles de endogamia, como es el caso de la localidad, Pioneros, Campeche,
México, FIS = 0.163, P < 0.05 (Delgado et al. 2011). No obstante, en la población
Caobas, Quintana Roo, México, no se encontraron niveles altos de endogamia, con
FIS = 0.057, P < 0.05 (Rebolledo
et al. 2018). Esto último puede ser debido a que la deficiencia de
individuos heterocigotos es poco común, dado que los pinos presentan
polinización cruzada. Según Delgado et al. (2011)
el hecho de que exista un patrón de baja DG y altos índices de endogamia en P.
caribaea var. hondurensis (FIS = 0.163) sugieren y
apoyan la hipótesis de la dinámica de metapoblación como lo han propuesto
previamente Jardón-Barbolla et al. (2011),
es decir que las poblaciones de esta especie están sujetas a factores de perturbación
como el fuego y tienen un alto recambio. Además, las poblaciones retienen
variantes alélicas ancestrales, probablemente por procesos de expansión,
mientras que otras poblaciones no los han retenido (contracción o extinción) (Jardón-Barbolla et al. 2011; Potter et al. 2015). El análisis de cuellos de
botella basado en modelos como el de exceso de heterocigosidad demuestran que a
pesar de usar marcadores altamente polimórficos como los SSRs, los niveles de
DG son bajos en P. caribaea var. hondurensis (He = 0.464)
con respecto a lo esperado para el equilibrio de deriva-mutación (Heq =
0.547) (Delgado et al. 2011), o P. taeda L.
(He = 0.64, Heq = 0.67) (Al-Rabab’ah
y Williams 2004). Por tanto, esto sugeriría que la pérdida de DG se debe a
la fragmentación de las poblaciones y a la disminución del Ne. Si este
último factor fuera el determinante en las estimaciones de la baja DG, sería
interesante cuantificar en qué punto del tiempo se observaron tales
contracciones del Ne (por ejemplo, Pleistoceno u Holoceno), dado que la
DG en Norteamérica ha sido moldeada en parte por las re-colonizaciones en
épocas glaciales. Otras hipótesis que se han planteado para explicar la baja DG
son los cuellos de botella durante las distintas etapas glaciales (Acosta et al. 2019) acompañadas de expansiones
rápidas (Marshall et al. 2002; Zinck y Rajora 2016). Por ejemplo, P.
maximartinezii con isoenzimas, presentó una DG baja (He = 0.122) e
índice de fijación relativamente alto (FST = 0.081), lo cual
indica deficiencia de heterocigotos (Ledig et al. 1999).
Algunas especies endémicas de México han demostrado declives demográficos, como
P. remota (Little) D.K. Bailey & Hawksw, donde se encontraron tres
poblaciones con déficit de DG (SSRn, He = 0.015-0.031) con base en el
modelo de exceso de heterocigosidad, lo cual sugiere eventos de cuellos de
botella que pudieron haber estado asociados al impacto del clima durante el
Pleistoceno (González et al. 2021).
Un proceso que incrementa la DG en las pináceas es la
hibridación, por ejemplo, en especies de Estados Unidos con marcadores RFLPs,
los índices de DG fueron mayores en las poblaciones híbridas (He =
0.67), con respecto a sus parentales, P. echinata (He = 0.554) y P.
taeda (He = 0.56) (Chen et al. 2004).
También en especies de México, donde las poblaciones híbridas de P.
montezumae Lamb. x P. pseudostrobus Lindl., distribuidas en la Faja
Volcánica Trans Mexicana (FVTM), mostraron mayores niveles de DG (He =
0.519) con respecto a sus parentales (P. montezumae, He = 0.409 y
P. pseudostrobus, He = 0.416). Además, se encontró una tasa de
introgresión mayor de las poblaciones de P. montezumae a las poblaciones
híbridas (Delgado et al. 2007). Otros estudios
abordan este tema, como la hibridación introgresiva de P. luzmariae Pérez
de la Rosa y P. herrerae Martínez (con marcadores moleculares AFLPs, Wehenkel et al. 2020) o P. monophylla Torr.
& Frém. y P. californiarum D.K. Bailey (con SNPs, Buck et al. 2020), así como eventos continuos de
introgresión en la zona híbrida de P. contorta Dougl. ex Loud var. latifolia
x P. banksiana Lamb (Burns et al. 2019).
Patrones de la diversidad genética en Pseudotsuga
Dentro de las especies reconocidas para este género de
Norteamérica (dos especies, Flora of North America, [utilizado el 10 de Julio, 2024], la
especie Pseudotsuga menziessi (Mirb.) Franco ha sido la que mayormente
se ha estudiado (Anexo, Tabla A4). No
encontramos información de estudios genéticos para P. macrocarpa (Vasey)
Mayr. Por tanto, sólo se ha documentado el 50% de la información de la genética
de poblaciones para este género. En México, las poblaciones del género Pseudotsuga,
presentan mayor DG dentro de las poblaciones (SSRcp, 72.2%) que entre
poblaciones (27.8%; Montiel et al. 2019),
tendencia que fue similar entre las variedades de P. menziesii con
isoenzimas, con 51% de la DG entre las variedades y 49% entre los subgrupos de
esta especie (Li y Adams 1989).
La especie P. menziesii tiene una distribución desde
Canadá a Estados Unidos (P. menziesii) y parte del norte de México (P.
menziesii var. glauca (Mayr) Franco), siendo México su distribución
más sureña. En este caso, Cruz-Nicolas et al.
(2011) encontraron la DG más baja en las poblaciones mexicanas (isoenzimas,
He = 0.021), mientras que en EU la DG fue más alta (isoenzimas, He =
0.178, Merkle y Adams 1987; He = 0.137,
Li y Adams 1989). La información ha sido
consistente con otros estudios dónde las poblaciones del norte de Estados
Unidos mostraron la mayor DG en la región costera, tanto con isoenzimas
(Heterocigosis total (HT) = 0.177), como con secuencias de
cloroplasto (He = 0.85-0.95, Nelson et al.
2003), mientras que las poblaciones del sur (México) tuvieron niveles
ligeramente más bajos (HT = 0.15) y una estructura genética
moderada (GST = 0.122) (Li y Adams
1989; Wei et al. 2011). Las diferencias de la DG
entre las poblaciones de Estados Unidos y México, podría ser el resultado de la
fragmentación de las poblaciones y, por tanto, de la reducción del flujo
génico.
El porcentaje del polimorfismo cuantificado en las
investigaciones fue variable en intervalos de 22.1% a 71.7%. Los bajos niveles
del polimorfismo resaltan en P. menziesii var. glauca con
distribución sureña (P = 22.1-28.3%, Li y Adams
1989; Cruz-Nicolás et al. 2011), mientras que el porcentaje del
polimorfismo en la var. menziesii distribuida al Norte de los Estados
Unidos fue mayor (P = 45.2-71.7%, Li y Adams 1989;
Merkle y Adams 1987; Schnabel et al. 1993). Estas diferencias podrían
ser resultado de la fragmentación de las poblaciones, promoviendo la pérdida de
variantes alélicas por deriva génica. De las dos variedades, la var. menziesii
tienen una distribución geográfica más amplia, por lo que es de esperarse
que contengan niveles del polimorfismo mayores con respecto a especies con una
distribución geográfica restringida (Haig 1998).
Los estudios filogeográficos indican que los altos niveles
de DG en P. menziesii se deben a que las zonas donde actualmente se
distribuye fueron recolonizadas a partir de uno o incluso más refugios
glaciales después del retroceso glacial (Pleistoceno) (Li
y Adams 1989). Este patrón es consistente con lo observado a través de
marcadores del cloroplasto, donde incluso los grupos distribuidos al norte son
los que albergan la mayor DG y de acuerdo con ello, se sugieren seis grupos
genéticos: tres de ellos de mayor DG, el P. menziesii costero del norte
(HS = 0.741), el del Sur (HS = 0.735) y el
norte de las montañas Rocallosas (HS = 0.75). El grupo de
transición de las montañas Rocallosas con valor intermedio de DG (HS =
0.676) y finalmente el grupo mexicano (P. menziesii var. glauca),
el cual tuvo la más baja DG (HS = 0.408) (Wei et al. 2011).
Así mismo, y al igual que otros géneros de pináceas, P.
menziesii está representada por variedades que hibridan en las zonas de
contacto, y varios estudios realizados con cloroplasto (Gugger et al. 2010, 2011), muestran información que sugiere la
existencia del intercambio e introgresión entre las poblaciones. En un estudio
de poblaciones de Estados Unidos y Canadá, se encontraron al menos ocho grupos
genéticos que corroboran estos eventos de introgresión (Van
et al. 2015), que concurren en tres posibles zonas híbridas con valores
similares de DG obtenidos con SSRn: el grupo I+III+IV+V, He = 0.88, el
grupo I+II, He = 0.86 y el grupo VI + VII, He = 0.87. Estas
estimaciones resultaron en valores de DG intermedios con los grupos donde no se
encuentran las mezclas anteriores (agrupamiento I (He = 0.92), II (He
= 0.82), III (He = 0.82), IV (He = 0.85), V (He =
0.9), VI (He = 0.9), VII (He = 0.8) y VIII (He = 0.8). De
hecho, el estudio previo de Gugger et al. (2010)
encontró la diversidad haplotípica más grande en Canadá (Hd = 0.592) con
respecto a las áreas geográficas no híbridas, distribuidas en la costa (Hd =
0.193), en las Rocallosas del norte (Hd = 0.413) y del sur (Hd =
0.520).
Análisis general de la diversidad genética (He)
vs. estructura genética (FST)
Dentro de los géneros incluidos en esta revisión,
encontramos una alta diversidad dentro de las poblaciones y baja estructura
genética. Esta última es mayor en las especies que tienen poblaciones pequeñas,
aisladas o fragmentadas. Por tanto, se esperaría encontrar una asociación
negativa entre la estructura genética y la DG (Delgado
y Piñero 2008); es decir, que en especies con mayor estructuración se
encontrarán menores valores de DG. Sin embargo, este patrón solo se observa con
el uso de los marcadores SSRs de núcleo (r = -0.06) y SNPs (r =
-0.70) en especies de pinos (Fig. 2, G, I),
aunque las correlaciones no fueron estadísticamente significativas (Tabla 1). Los resultados de tales asociaciones se
explicarían en parte por las características biológicas de las especies, que
poseen tamaños efectivos poblacionales grandes y son de amplia distribución o
localmente abundantes, con mecanismos eficientes de dispersión del polen por el
viento y su fecundación cruzada. Las demás correlaciones (r de Pearson)
de los otros géneros resultaron positivas y sin significancia estadística, a
excepción de los datos de Abies y Picea con isoenzimas, que
fueron estadísticamente significativos (Fig. 2).
La información de las especies de los géneros de Pinaceae evaluadas en las
correlaciones de Pearson se encuentra en la Tabla 5 del
Anexo.
Adicionalmente, en esta investigación analizamos la relación
entre el estimador (He) de la diversidad genética, como variable de
respuesta, y las variables especie y marcador como variables fijas, usando
modelos lineales generalizados (GLM, por sus siglas en inglés) con función de
enlace logit y distribución de errores binomial. Los resultados sugieren que no
hay diferencias estadísticamente significativas (Tabla 2).
Asimismo, el análisis de varianza (ANOVA) a partir de los resultados obtenidos
del GLM sugiere que no hay una relación de las variables fijas y la variable de
respuesta (Tabla 3). Finalmente, calculamos la
proporción de la relación entre la desviación explicada por el modelo y la
desviación total, utilizando los componentes de la desviación nula y la del
modelo GLM, con el fin de calcular la R2 (coeficiente de
determinación) del modelo GLM obtenido, el cual fue de 0.89, lo que confirma la
ausencia de relación de las variables y, por tanto, de que la especie o el
marcador expliquen la diversidad genética.
Tabla 1. Resultados
de las correlaciones de Pearson (r) para los distintos géneros de Pinaceae
revisados en este estudio.
Table
1. Results of Pearson correlations (r) for the different Pinaceae genera reviewed in this study.
t
es el valor estadístico de la prueba t, df son los grados de libertad; r es la
estimación del coeficiente de correlación de Pearson; y el valor p es el nivel
de significancia de la prueba t (95%).
t is the
statistical value of the t-test; df are the degrees of freedom; r is the
estimate of the Pearson correlation coefficient; and p-value is the
significance level of the t-test (95%).
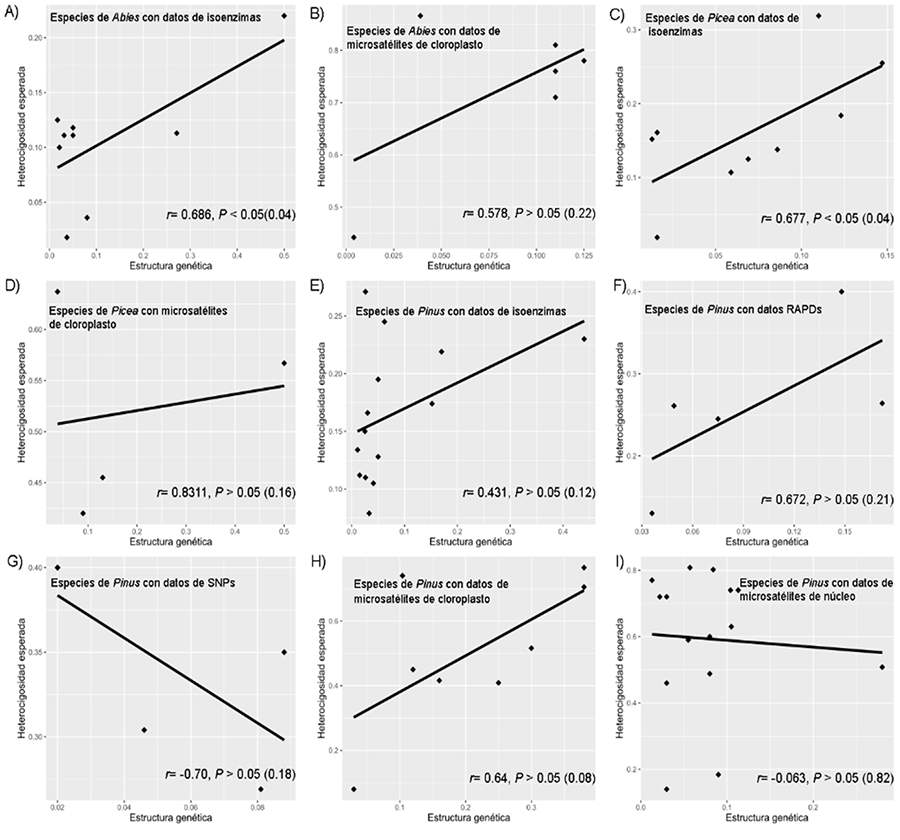
Figura 2. Correlaciones
de Pearson entre la diversidad genética (heterocigosis esperada, He) y la
estructura genética (FST). Datos obtenidos con isoenzimas (A)
y SSRcp (B) en especies de Abies; con isoenzimas (C) y
SSRcp (D) en especies de Picea; y en Pinus con isoenzimas
(E), RAPDs (F), SNPs (G), SSRcp (H) y SSRn (I).
Las correlaciones fueron estimadas con los datos obtenidos de diversas fuentes
que han estimado la diversidad genética en la familia Pinaceae. La información sobre
las especies evaluadas se encuentra en la Tabla 5 del
Anexo.
Figure 2. Pearson correlations between genetic diversity (expected heterozygosis, He) and genetic structure (FST). Data obtained with isoenzymes (A) and SSRcp (B) in Abies species; with isoenzymes (C) and SSRcp (D) in Picea species; and in Pinus with isoenzymes (E), RAPDs (F), SNPs (G), SSRcp (H) and SSRn (I). Correlations were estimated with data obtained from various sources
that have estimated genetic diversity in the Pinaceae family. Information on
the species assessed is given in Table 5 of the Appendix.
Tabla 2. Resultados de los
estimadores del coeficiente beta y sus niveles de significancia del modelo
lineal generalizado (GLM, por sus siglas en inglés), para las variables fijas
especie y marcador molecular, tomando como variable de respuesta la diversidad
genética medida como heterocigosidad esperada (He).
Table
2. Results of the beta coefficient estimators and
their significance levels of the generalized linear model (GLM), using species
and molecular marker as fixed variables, and as response variable to genetic
diversity measured as expected heterozygosity (He).
Tabla 3. Anova del
modelo lineal generalizado (GLM) presentado en la Tabla 2.
Table
3. Anova of the generalized linear model (GLM) used
in Table 2.
Conclusiones
La revisión de estudios genéticos en la familia Pinaceae de
Norteamérica muestra diferencias en las estimaciones de la DG con diferentes
marcadores moleculares; sin embargo, hay congruencia en las inferencias de la
historia evolutiva de las diferentes especies. Encontramos que la mayor DG se
encuentra dentro de las poblaciones y es más baja entre las poblaciones,
independientemente del tipo de marcador molecular utilizado (dominante o
codominante). Es decir, que el sistema de cruce de las especies estudiadas
permite el mantenimiento de altos niveles de diversidad y poca diferenciación
poblacional. No obstante, los SSRs mostraron mayores niveles de DG, lo cual se
atribuye a las altas tasas de mutación de estas regiones. La DG encontrada en
las poblaciones de cada una de las especies es explicada por diversos procesos
evolutivos históricos y contemporáneos, como la deriva génica, endogamia,
recolonizaciones post-glaciales y flujo génico. Los modelos GLM indican que las
variables especie o marcador molecular tomados como factores fijos en el
análisis, no tienen ninguna relación con la DG (variable tomada como factor de
respuesta) para las especies revisadas en este estudio. Los estudios revisados
también demuestran que la hibridación es un proceso evolutivo de importancia en
estos grupos de árboles, dado que poblaciones híbridas presentan niveles más
altos de DG, lo cual puede servir de materia prima para la evolución y origen
de nuevas especies. Los resultados de esta revisión muestran que el
conocimiento sobre la diversidad genética en las Pináceas de Norte América
avanza, aunque es necesario realizar más estudios de genética de poblaciones
para los cuatro géneros, con mayor énfasis en Pinus. Consideramos que es
prioritario conservar “in situ” aquellas poblaciones que son relictas de
las glaciaciones, de distribución restringida o endémicas, ante la devastación
antrópica y el cambio climático. Finalmente, los nuevos métodos de
secuenciación masiva que permiten obtener una enorme cantidad de datos
genómicos para detectar variantes genómicas (por ejemplo, SNPs) deberían
permitirnos tener cuantificaciones más finas sobre los parámetros de la
genética de poblaciones, resultados más claros sobre la estructura de las
poblaciones y la propuesta de nuevas hipótesis que nos permitan entender la
historia evolutiva de las especies de Norte América.
Disponibilidad de los datos
Este artículo no utiliza conjuntos de datos.
Contribución de los autores
Alfredo Ortiz-Martínez: Conceptualización, Investigación,
Redacción - borrador original, Redacción – revisión y edición, Análisis formal,
Metodología, Curaduría de datos, Visualización y Supervisión. Alejandra
Moreno-Letelier: Conceptualización, Redacción - revisión y edición,
Visualización y Supervisión. Patricia Delgado Valerio: Conceptualización,
Metodología, Redacción -borrador original, Redacción – revisión y edición,
Visualización y Supervisión; Daniel Piñero: Redacción – borrador original,
Visualización, Investigación. Carlos Ortiz-Martínez: Investigación, Redacción
-revisión y edición, Metodología, Curaduría de datos, Análisis formal.
Alejandra Vázquez Lobo: Conceptualización, Redacción – revisión y edición,
Análisis formal, Metodología, Visualización y Supervisión.
Conflictos de interés
Los autores declaran no tener ningún conflicto de interés,
financiero o personal, en la información, presentación de los datos y los
resultados de este artículo.
Agradecimientos
Al programa del Doctorado del Posgrado en Ciencias
Biológicas, UNAM. A CONAHCyT por el apoyo de la beca doctoral (CVU: 706348). Al
Dr. David S. Gernandt por proporcionar las imágenes de las especies incluidas
en el mapa de distribución de las Pináceas de Norteamérica. Finalmente, a cada
uno de los autores que se encuentran citados en este artículo de revisión, ya
que, sin sus investigaciones, esto no sería posible.
Referencias
Aagaard, J.E., Krutovskii, K.V.,
Strauss, S.H. 1998. RAPDs and allozymes exhibit similar
levels of diversity and differentiation among populations and races of
Douglas-fir. Heredity 81, 69-78.
Acosta, J., Fahrenkrog, A.M., Neves, L.G., Resende, F.R., Dervinis, C.,
Davis, J.M., Holliday, J.A. et al. 2019. Exome resequencing reveals
evolutionary history, genomic diversity, and targets of selection in the
conifers Pinus taeda and Pinus elliottii. Genome
Biology and Evolution 11(2), 508-520.
Aguirre-Limón, V.,
Alanís-Flores, G., González-Rojas, J.I., Flores-Suárez, A., Favela-Lara, S.
2017. Variación genética de Pinus pinceana Gordon evidencia de
conectividad en poblaciones fragmentadas. Revista Mexicana de Ciencias
Forestales 8(43), 39-63.
Aguirre-Planter, E.,
Furnier, G.R., Eguiarte, L.E. 2000. Low levels of genetic
variation within and high levels of genetic differentiation among populations
of species of Abies from southern Mexico and Guatemala. American
Journal of Botany 87(3), 362-371.
Al-Rabab’ah,
M.A., Williams, C.G. 2004. An ancient bottleneck in
the Lost pines of central Texas. Molecular Ecology 13(5), 1075-1084.
Arzate-Fernández,
F., Gutiérrez-González, G., Heredia-Bobadilla, R.L. 2016. Diversidad
genética de dos especies de coníferas en el Nevado de Toluca: una alternativa
de conservación. 136 p., Universidad Autónoma del Estado de México. Toluca,
México.
Bashalhanov, S.,
Johnson, J.S., Rajora, O.P. 2023. Postglacial phylogeography,
admixture, and evolution of red spruce (Picea rubens Sarg.) in Eastern
North America. Frontiers in Plant Science 14, 1272362
Boys, J., Cherry, M., Dayanandan, S. 2005. Microsatellite analysis
reveals genetically distinct populations of red pine (Pinus resinosa,
Pinaceae). American Journal of Botany 92, 833-841.
Bruederle, L.P., Tomback, D.F., Kelly, K.K., Hardwick, R.C. 1998. Population
genetic structure in a bird-dispersed pine, Pinus albicaulis (Pinaceae).
Canadian Journal of Botany 76, 83-90.
Buck, R., Hyasat, S., Hossfeld, A., Flores-Rentería, L. 2020. Patterns
of hybridization and cryptic introgression among one- and four-needled pinyon
pines. Annals of Botany 126(3), 401-411.
Burns, I., James, P.M.A., Coltman, D.W., Cullingham, C.I. 2019. Spatial
and genetic structure of the lodgepole x jack pine hybrid zone. Canadian
Journal of Forest Research 49, 844-853.
Byrne, T., Farrelly, N., Kelleher, C., Hodkinson, T.R., Byrne, S.L.,
Barth, S. 2022. Genetic diversity and structure of a diverse population of Picea
sitchensis using Genotyping-by-Sequencing. Forests 13(9),
1511.
Castro-Félix, P., Pérez
de la Rosa, J.A., Vargas, A.G., Velásquez, M.S., Santerre, A., López-Dellamary,
T.F., Villalobos-Arámbula, A.R. 2008. Genetic relationships
among Mexican white pines (Pinus, Pinaceae) based on RAPD markers. Biochemical
Systematics and Ecology 36(7), 523-530.
Chen, J., Tauer, C.G., Bai, G., Huang, Y., Payton, M.E., Holley, A.G.
2004. Bidirectional introgression between Pinus taeda and Pinus
echinata: Evidence from morphological and molecular data. Canadian
Journal of Forest Research 34(12), 2508-2516.
Cinget, B., Gérardi, S., Beaulieu, J., Bousquet, J. 2015. Less pollen
mediated gene flow for more signatures of glacial lineages: congruent evidence
from balsam fir cpDNA and mtDNA for multiple refugia in eastern and central
North America. PLoS ONE 10(4), e0122815.
Clark, C.M., Wentworth, T.R., O’Malley, D.M. 2000. Genetic discontinuity
revealed by chloroplast microsatellites in eastern North American Abies
(Pinaceae). American Journal of Botany 87(6), 774-782.
Cruz-Nicolás, J., Vargas-Hernández, J.J., Ramírez-Vallejo, P., López-Upton, J.
2011. Genetic diversity and differentiation of Pseudotsuga
menziesii (Mirb.) Franco populations in México. Revista
Fitotecnia Mexicana 34(4), 233-240
Cruz-Nicolás, J.,
Giles-Pérez, G., González-Linares, E., Múgica-Gallart, J., Lira-Noriega, A.,
Gernandt, D.S., Eguiarte, E.L., et al. 2019. Contrasting
evolutionary processes drive morphological and genetic differentiation in a
subtropical fir (Abies, Pinaceae) species complex. Botanical Journal
of the Linnean Society 192(2), 401-420.
Davidson,
R., El-Kassaby, Y.A. 1997. Genetic diversity and
gene conservation of Pacific Silver fir (Abies amabilis) on Vancouver
Island, British Columbia. Forest Genetics 4(2), 85-98.
De La Torre, A.R., Lin,
Y-C., de Peer, Y.V., Ingvarsson, P.K. 2015. Genome-wide
analysis reveals diverged patterns of codon bias, gene expression, and rates of
sequence evolution in Picea gene families. Genome Biology and
Evolution 7(4), 1002-1015.
Decker, S.A. 2022. Families in the Sky: investigating the population structure of
Pinus longaeva. MasterThesis. Brigham Young University, USA.
Delgado, P., Piñero, D. 2008.
Marcadores moleculares, variación genética y filogeografía en árboles
forestales. En: Sánchez, V. (Ed.), Ecología, Manejo y conservación de los
ecosistemas de montaña en México, pp. 1-19. Editorial MundiPrensa México,
S. A. de C.V. México, D.F, México.
Delgado, P., Piñero, D.,
Chaos, A., Pérez-Nasser, N., Álvarez-Buylla, E.R. 1999. High
population differentiation and genetic variation in the endangered Mexican pine
Pinus rzedowskii (Pinaceae). American Journal of Botany
86(5), 669-676.
Delgado, P., Salas-Lizana,
R., Vázquez-Lobo, A., Wegier, A., Anzidei, M., Álvarez-Buylla, E.R., Vendramin,
G.G., et. al. 2007. Introgressive hybridization in Pinus
montezumae Lamb. and P. pseudostrobus Lindl. (Pinaceae):
morphological and molecular (cpSSR) evidence. International Journal of Plant
Sciences 168(6), 861-875.
Delgado, P., Piñero, D.,
Rebolledo, V., Jardón, L., Chi, F. 2011. Genetic variation and
demographic contraction of the remnant populations of Mexican Caribbean pine (Pinus
caribaea var. hondurensis: Pinaceae). Annals
of Forest Science 68, 121-128.
Dvorak, W.S., Potter, K.M., Hipkins, V.D., Hodge, G.R. 2009. Genetic
diversity and gene Exchange in Pinus oocarpa, a mesoamerican pine with
resistance to the pitch canker fungus (Fusarium circinatum). International
Journal of Plant Sciences 170(5),
609-626.
Echt, C.S., DeVerno, L.L., Anzidei, M., Vendramin, G.G. 1998. Chloroplast microsatellites reveal population genetic diversity in
red pine, Pinus resinosa Ait. Molecular Ecology 7,
307-316.
Eckert, A.J., Eckert, M.L., Hall, B.D. 2010. Effects of historical
demography and ecological context on spatial patterns of genetic diversity
within foxtail pine (Pinus balfouriana; Pinaceae) stands located in the
Klamath Mountains, California. American Journal of Botany 97(4),
650-659.
Edwards,
M.A., Hamrick, J.L. 1995. Genetic variation in
shortleaf pine, Pinus echinata Mill. (Pinaceae). Forest Genetics
2(1), 21-28.
El-Kassaby,
Y.A., Ritland, K. 1996. Genetic variation in low
elevation Douglas-fir of Columbia Britanica and its relevance to gene
conservation. Biodiversity and Conservation 5, 779-794.
Epperson, B.K., Telewski, F.W., Willyard, A. 2009. Chloroplast diversity in
a putative hybrid swarm of Ponderosae (Pinaceae). American Journal of Botany
96(3), 707-712.
Ettl, B.J.,
Peterson, D.J. 2001. Genetic variation of subalpine
fir (Abies lasiocarpa (Hook.) Nutt.) in the Olympic Mountains, Wa, USA. Silvae
Genetica 50(3), 145-153.
Fageria,
M.S., Rajora, O.P. 2013. Effects of harvesting of
increasing intensities on genetic diversity and population structure of white
spruce. Evolutionary Applications 6(5), 778-794.
Farjon, A.,
Filer, D. 2013. Atlas of the World’s Conifers: an
analysis of their distribution, biogeography, diversity and conservation
status. Brill (Ed.), Boston, USA.
Favela, L.S. 2010. Population variation in the endemic Pinus culminicola
detected by RAPD. Polibotanica 30, 55-67.
Flora
of North America. 1993. Pinaceae.
[utilizado el 10/06/2024]. Disponible en:http://www.efloras.org/florataxon.aspx?flora_id=1&taxon_id=10691
Fuentes-Amaro, S.L.,
Legaria-Solano, J.P., Ramirez-Herrera, C. 2019. Estructura genética de
poblaciones de Pinus cembroides de la región central de México. Revista
Fitotecnia Mexicana 42(1), 57-65.
Gamache, I.,
Jaramillo-Correa, J.P., Payette, S., Bousquet, J. 2003. Diverging
patterns of mitochondrial and nuclear DNA diversity in subarctic black spruce:
imprint of a founder effect associated with postglacial colonization. Molecular
Ecology 12(4), 891-901.
Gapare, W. J., Aitken, S.N., Ritland, C.E. 2005. Genetic diversity of core
and peripheral Sitka spruce (Picea sitchensis (Bong.) Carr) populations:
implications for conservation of widespread species. Biological Conservation
123(1), 123-123.
García-Gómez, G.M.R., Floran, V., Östlund, L., Mullin, T.J.T., Andersson, G.B.
2015. Genetic diversity and inbreeding in natural and managed populations of
scots pine. Tree Genetics & Genomes 11,
28.
Godbout, J., Beaulieu, J., Bousquet, J. 2011. Phylogeographic structure of
Jack pine (Pinus banksiana: Pinaceae) supports the existence of a
coastal glacial refugium in Northeastern North America. American Journal of
Botany 97(11), 1903-1912.
Godbout, J., Yeh, F.C., Bousquet, J. 2012. Large-scale asymmetric
introgression of cytoplasmic DNA reveals Holocene range displacement in a North
American boreal pine complex. Ecology and Evolution 2(8),
1853-1866.
Gernandt, D.S., Pérez-de
la Rosa, J.A. 2014. Biodiversidad de Pinophyta (coníferas) en México. Revista Mexicana de Biodiversidad85, 126-133.
Godt, M.J.W., Hamrick, J.L., Edwards-Burke, M.A., Williams, J.H. 2001.
Comparisons of genetic diversity in white spruce (Picea glauca) and Jack
pine (Pinus banksiana) seed orchards with natural populations. Canadian
Journal of Forest Research 31(6):943-949
González, J.A., Delgado, P., Molina, A., Flores, C., Vargas, J.P., García,
J.J. 2021. Diversidad genética en poblaciones de Pinus remota
(Pinaceae) del noroeste de México: evidencias de declinamiento demográfico
histórico. Acta Botánica Mexicana 128, e1890.
Gugger, P.F., Sugita, S.,
Cavender-Bares, J. 2010. Phylogeography of Douglas‐fir
based on mitochondrial and chloroplast DNA sequences: testing hypotheses from
the fossil record. Molecular Ecology 19(9), 1877-1897.
Gugger, P.F.,
Gonzalez-Rodríguez, A., Rodríguez-Correa, H., Sugita, S., Cavender-Bares, J.
2011. Southward Pleistocene migration of Douglas-fir into
Mexico: phylogeography, ecological niche modeling, and conservation of ‘rear
edge’ populations. New Phytologist 189(4), 1185-1199.
Haig, S.M. 1998. Molecular contributions to conservation. Ecology 79, 413-425.
Hamilton,
J.A., Aitken, S.N. 2013. Genetic and morphological
structure of a spruce hybrid (Picea sitchensis x P. glauca) zone
along climatic gradient. American Journal of Botany 100(8), 1651-1662.
Hamrick,
J.L., Schnabel, A.F., Wells, P.V. 1994. Distribution of genetic diversity
within and among populations of Great Basin conifers. En: Harper, K.T., St.
Clair, L.L., Thorne, K.H., Hess, W.W. (eds.), Natural history of the
Colorado Plateau and Great Basin. pp. 31-61. University Press of Colorado,
Niwot, Colorado, USA.
Hansen, O.K., Lamour, K., Whetten, R., Xu, J., Cuenca, A., Islam, Md.S.,
Nielsen, U.B. 2020. Ad hoc breeding of a genetically depauperate landrace of
noble fir (Abies procera Rehder) using SNP genotyping via
high-throughput targeted sequencing. Tree Genetic & Genomes 16(5), 63.
Haselhorst,
M., Buerkle, C.A. 2013. Population genetic
structure of Picea engelmannii, P. glauca and their previously
unrecognized hybrids in the central Rocky Monutains. Tree Genetics &
Genomes 9:669-681.
Haselhorst, M.S.H., Parchman, T.L., Buerkle, C.A. 2019. Genetic evidence for
species cohesiion, substructure and hybrids in spruce. Molecular Ecology
28(8), 2029-2045.
Hendrickson,
B., Anderson, M.R., Nelson, C.D., Jossernd, S., Echt, C., Berkman, L.K.,
Koppelman, J.B. 2018. Genetic diversity and population structure of shortleaf
pinne (Pinus echinata) in the Missouri Ozarks. The American Midland
Naturalist Journal 180, 37-51.
Ivetić, V., Devetaković, J. 2017. Concerns and evidence on genetic diversity in
planted forests. Reforesta 3, 196-207.
Jaramillo-Correa, J.P., Beaulieu, J., Ledig, F.T., Bousquet, J. 2006. Decoupled
mitochondrial and chloroplast DNA population structure reveals Holocene
collapse and population isolation in a threatened Mexican-endemic conifer. Molecular
Ecology 15(10), 2787-2800.
Jaramillo-Correa,
J.P., Aguirre-Planter, E., Khasa, D.P., Eguiarte, L.E., Piñero, D., Furnier,
G.R., Bousquet, J. 2008. Ancestry and divergence of
subtropical montane forest isolates: molecular biogeography of the genus Abies
(Pinaceae) in southern México and Guatemala. Molecular Ecology
17(10), 2476-2490
Jardón-Barbolla, L.,
Delgado-Valerio, P., Geada-López, G., Vázquez-Lobo, A., Piñero, D. 2011. Phylogeography of Pinus subsection Australes in the
Caribbean Basin. Annals of Botany 107(2), 229-241.
Jorgensen, S., Hamrick, J.L., Wells, P.V. 2002. Regional patterns of genetic
diversity in Pinus flexilis (Pinaceae) reveal complex species history. American
Journal of Botany 89(5), 792-800.
Kim, M-S., Richardson, B.A., McDonald, G.I., Klopfenstein, N.B. 2010.
Genetic diversity and structure of western white pine (Pinus monticola)
in North America: a baseline study for conservation, restoration, and
addressing impacts of climate change. Tree Genetics & Genomes 7(1), 11-21.
Konnert, M.,
Ruetz, W.F. 1997. Genetic variation among
provenances of Abies grandis from the pacific Northwest. Forest
Genetics 4(2), 77-84.
Krutovsky,
K.V., St. Clair, J.B., Saich, R., Hipkins, V.D., Neale, D.B. 2009. Estimation
of population structure in coastal Douglas-fir [Pseudotsuga menziesii
(Mirb.) Franco var. menziesii] using allozyme and microsatellite
markers. Tree Genetics and Genomes 5, 641–658.
Lea, M.V., Syring, J., Jennings, T., Cronn, R., Bruederle, L.P., Neale,
J.R., Tomback, D.F. 2018. Development of nuclear microsatellite loci for Pinus
albicaulis Engelm. (Pinaceae), a conifer of conservation concern. PLoS
ONE 13(10), e0205423.
Ledig, F.T., Jacob-Cervantes, V., Hodgskiss, P.D., Eguiluz-Piedra, T.
1997. Recent evolution and divergence among populations of a rare Mexican
endemic, Chihuahua spruce, following Holocene climatic warming. Evolution
51(6), 1815-1827.
Ledig, F.T., Conkle, M.T., Bermejo-Velázquez, B., Eguiluz-Piedra, T., Hodskiss,
P.D., Johnson, D.R., Dvorak, W.S. 1999. Evidence for an extreme bottleneck in a
rare mexican pinyon: genetic diversiry, disequilibrium, and the matyng system
in Pinus maximartinezii. Evolution 53(1), 91-99.
Ledig, F.T., Capó, M.A., Hodgskiss, P.D., Sbay, H., Flores, L.C.,
Thompson, C.M., Bermejo, V.B. 2001. Genetic diversity and the mating system of
a rare mexican piñon, Pinus pinceana, and a comparison with Pinus
maximartinezii (Pinaceae). American Journal of Botany 88,
1977-1987.
Ledig, F.T., Hodgskiss, P.D., Jacob-Cervantes, V. 2002. Genetic
diversity, mating system, and conservation of a Mexican subalpine relict, Picea
mexicana Martínez. Conservation Genetics 3(2), 113-122.
Ledig, F.T., Hodgskiss, P.D., Johnson, D.R. 2005. Genic diversity,
genetic structure, and mating system of Brewer spruce (Pinaceae), a relict of
the Arcto-Tertiary forest. American Journal of Botany 92(12), 1975-1986.
Ledig, F.T., Hodgskiss, P.D., Johnson, D.R. 2006. The structure of
genetic diversity in Engelmann spruce and a comparison with blue spruce. Canadian
Journal of Botany 84(12), 1806-1828.
Lee, S.W., Ledig, F.T., Johnson, D.R. 2002. Genetic variation at
allozyme and RAPD markers in Pinus longaeva (Pinaceae) of the white
mountains, California. American Journal of Botany 89(4), 566-577.
Li, P., Adams,
W.T. 1989. Range-wide patterns of allozyme
variation in Douglas-fir (Pseudotsuga menziesii). Canadian Journal of
Forest Research 19, 149-161.
Liu, J.J., Sniezko, R., Murray, M., Wabg, N., Chen, H., Zamany, A.,
Sturrock, R. N., Savin, D., Kegley, A. 2016. Genetic diversity and population
structure of whitebark pine (Pinus albicaulis Engelm.) in Western North
America. PLoS ONE 11(12), e0167986.
Mahalovich,
M.F., Hipkins, V.D. 2011. Molecular genetic
variation in whitebark pine (Pinus albicaulis Engelm.) in the Inland
West. En: Keane, R.E., Tomback, D.F., Murray, M.P., Smith, C.M. (Eds.), The
future of high-elevation, five-needle white pines in Western North America:
Proceedings of the High Five Symposium. 28-30 June 2010; Missoula, MT. (Proceedings
RMRS-P-63). Fort Collins, CO: U.S. Department of Agriculture, Forest Service,
Rocky Mountain Research Station, USA. 376 p.
Mallet, J. 2008. Hybridization, ecological races and the nature of species:
empirical evidence for the ease of speciation. Philosophical Transactions of
the Royal Society B 363(1506), 2971-2986.
Marquardt, P.E., Echt, C.S., Epperson, B.K., Pubanz, D.M. 2007. Genetic
structure, diversity, and inbreeding of eastern white pine under different
management conditions. Canadian Journal of Forest Research 37(12), 2652-2662.
Marshall, H.D., Newton, C., Ritland, K. 2002. Chloroplast phylogeography and
evolution of highly polymorphic microsatellites in lodgepole pine (Pinus
contorta). Theoretical and Applied Genetics 104(2-3), 367-378.
Mehes, M., NKongolo, K.K., Michael, P. 2009. Assessing genetic diversity
and structure of fragmented populations of eastern white pine (Pinus strobus)
and western white pine (P. monticola) for conservation management. Journal of Plant Ecology 2(3), 143-151.
Mendoza-Maya, E.,
Espino-Espino, J., Quiñones-Pérez, C.Z., Flores-López, C., Wehenkel, C.,
Vargas-Hernández, J.J., Sáenz-Romero, C. 2015. Propuesta de conservación de
tres especies mexicanas de Picea en peligro de extinción. Revista Fitotecnia Mexicana 38(3),
235-247.
Merkle, S.A.,
Adams, W.T. 1987. Patterns of allozyme variation
within and among Douglas-fir breeding zones in Southwest Oregon. The Canadian
Journal of Forest Research 17(5), 402-407.
Montiel, C.P., Cortés-Cruz, M., Mendoza-Carrillo, M.C., Cruz-Izquierdo, S.,
López-Upton, J., Sandoval, P.I., Guerra, C.V. 2019. Diversity and genetic
structure inferred with microsatellites in natural populations of Pseudotsuga
menziesii (Mirb.) Franco (Pinaceae) in the central region of Mexico.
Forests 10(2),
101.
Morales-Nieto, C.R.,
Siqueiros-Candia, M., Álvarez-Holguín, A., Corrales-Lerma, R.,
Alarcón-Bustamante, M., Martínez-Salvador, M. 2021. Estructura y diversidad
genética en poblaciones de Pinus engelmannii Carr. en Chihuahua, México.
Revista Fitotecnia Mexicana 43,
2.
Moran, G.F., Adams, W.T. 1989. Microgeographical patterns of allozyme differentiation in
Douglas-Fir from southwest Oregon. Forest Science 35(1), 3-15.
Myers, E.R., Chung, M.Y., Chung, M.G. 2007. Genetic diversity and spatial
genetic structure of Pinus strobus (Pinaceae) across an island landscape
inferred from allozyme and cpDNA markers. Plant Systematics and Evolution
264(1-2), 15-30.
Nadeau, S., Godbout, J., Lamothe, M., Gros-Louis, M.C., Isabel, N.,
Ritland, K. 2015. Contrasting patterns of genetic diversity across the ranges
of Pinus monticola and P. strobus: a comparison between Eastern
and Western North American postglacial colonization histories. American
Journal of Botany 102(8), 1342-1355.
Narendrula,
R. Nkongolo, K.K. 2012. Genetic variation in Picea
mariana x P. rubens hybrid populations assessed with ISSR and RAPD
markers. American Journal of Plant Sciences 3, 731-737.
Nelson,
R.J., Stoehr, M., Cooper, G., Smith, C., Mehl, H. 2003. High levels of
chloroplast genetic variation differentiate coastal and interior Douglas-Fir (Pseudotsuga
menziesii) lineages in southern British Columbia. Forest Genetics 10(2), 153-157.
Newton, A.C., Allnut, T.R., Dvorak, W.S., Del Castillo, R.F., Ennos, R.A.
2002. Patterns of genetic variation in Pinus chiapensis, a threatened
Mexican pine, detected by RAPD and mitochondrial DNA RFLP markers. Heredity 89, 191-198.
Niebling,
A.U., Conkle, M.T. 1990. Diversity of washoe pine
and comparisons with allozymes of ponderosa pine races. Canadian Journal of
Forest Research 20(3), 298-308.
Olmstead, R.G., Palmer, J.D. 1994.
Chloroplast DNA systematics: a review of methods and data analysis. American
Journal of Botany 81(9), 1205-1224.
Ortíz-Medrano, A., Moreno-Letelier, A., Piñero, D. 2008. Fragmentación y
expansión demográfica en las poblaciones mexicanas de Pinus ayacahuite
var. ayacahuite. Boletín de la Sociedad Botánica de México 83, 25-36.
Parchman, T.L., Geits,
K.S., Grahnen, J.A., Benkman, C.W., Buerkle, C.A. 2011. Transcriptome
sequencing in an ecologically important tree species: assembly, annotation, and
marker Discovery. BMC Genomics 11, 180.
Parker,
K.C., Hamrick, J.L., Parker, A.J., Stacy, E.A. 1997. Allozyme diversity in Pinus
virginiana (Pinaceae): intraspecific and interspecific comparisons. American
Journal of Botany 84(10), 1372-1382.
Parker,
K.C., Hamrick, J.L., Parker, A.J., Nason, J.D. 2001. Fine-scale genetic
structure in Pinus clausa (Pinaceae) populations: effects of disturbance
history. Heredity 87, 99-113<.
Potter,
K.M., Frampton, J., Josserand, S.A., Nelson, D. 2008. Genetic variation and
population structure in Fraser Fir (Abies fraseri): a microsatellite
assessment of young trees. Canadian Journal of Forest Research 38,
2128-2137.
Potter, K.M., Hipkins, V.D., Mahalovich, M.F., Means, R.E. 2015. Nuclear
genetic variation across the range of ponderosa pine (Pinus ponderosa):
Phylogeographic, taxonomic, and conservation implications. Tree
Genetics & Genomes 11, 38-60.
Quiñones-Pérez, C.Z.,
González-Elizondo, M.S., Wehenkel, C. 2017. Ruling out genetic
erosion in Picea chihuahuana Martínez. New Forest 48(2), 201-215.
Raja, R.G., Tauer, C., Wittwer, G.R.F., Huang, Y.
1997. Isoenzyme variation and genetic structure in natural populations of
shortleaf pine (Pinus echinata). Canadian Journal of Forest Research
27(5), 740-749.
Rajora,
O.P., Dancik, B.P. 2000. Population genetic
variation, structure, and evolution in Engelmann spruce, white spruce, and
their natural hybrid complex in Alberta. Canadian Journal of Botany
78(6), 768-780.
Rajora,
O.P., Pluhar, S.A. 2003. Genetic
diversity impacts of forest fires, forest harvesting, and alternative
reforestation practices in black spruce (Picea mariana). Theorical
and Applied Genetics 106(7), 1203-1212.
Rajora,
O.P., DeVerno, L., Mosseler, A., Innes, D.J. 1998. Genetic diversity and
population structure of disjunct Newfoundland and central Ontario populations
of eastern white pine (Pinus strobus). Canadian Journal of Botany 76,
500-508
Rajora, O.P., Mann, I. K., Shi, Y-Z. 2005. Genetic
diversity and population structure of boreal white spruce (Picea glauca)
in pristine conifer-dominated and mixedwood forest stands. Canadian Journal
of Botany 83(9), 1096-1105.
Rajora,
O.P., Eckert, A.J., Zinck, J.W.R. 2016. Single-locus versus multilocus patterns
of local adaptation to climate in eastern white pine (Pinus strobus,
Pinaceae). PLoS ONE 11(7), e0158691.
Rebolledo, C.V., Jardón,
B.L., Ramírez, M.I., Vázquez-Lobo, A., Piñero, D., Delgado, P. 2018. Genetic variation and dispersal patterns in three varieties of Pinus
caribaea (Pinaceae) in the Carribbean Basin. Plant Ecology and Evolution
151(1), 61-76.
Ritland,
K., Miscampbell, A., Cartwriht, C., Bohlmann, N., Ritland, C. 2022. Subalpine
fir microsatellite variation reveals the complex relationships between var. lasiocarpa
and var. bifolia. Canadian Journal of Forest Research 52(6), 901-909.
Sáenz-Romero,
C., Tapia-Olivares, B.L. 2003. Pinus oocarpa
isoenzymatic variation along an altitudinal gradient in Michoacán, México. Silvae
Genetica 52(5), 237-240.
Schnabel, A., Hamrick, J.L., Well, P.V. 1993. Influence of Quaternary
history on the population genetic structure of Douglas-fir (Pseudotsuga
menziesii) in the Great Basin. Canadian Journal of Forest Research 23(9), 1900-1906.
Schmidtling,
R.C., Hipkins, V. 1998. Genetic diversity in
longleaf pine (Pinus palustris): influence of historical and
prehistorical events. Canadian Journal of Forest Research 28:1135-1145.
Shea, K.L. 1989. Genetic variation between and within populations of Engelmann
spruce and subalpine fir. Genome 33(1), 1-8.
Shea, K.L.,
Furnier, G.R. 2002. Genetic variation and
population structure in central and isolated populations of balsam fir, Abies
balsamea (Pinaceae). American Journal of Botany 89(5), 783-791.
Smith, D.R. 2016. The past, present and future of mitochondrial genomics: have we
sequenced enough mtDNAs? Briefings in Functional Genomics 15(1), 47-54.
Soltis,
P.S., Soltis, D.E. 2009. The role of hybridization
in plant speciation. Annual Review of Plant Biology 60, 561-581.
Tremblay,
M., Simon, J.P. 1989. Genetic structure of marginal
populations of white spruce as its northern limit of distribution in
Nouveau-Quebec. Canadian Journal of Forest Research 19(11), 1371-1379.
Tsutsui, K., Suwa, A., Sawada, K., Kato, T., Ohsawa, T.A., Watano, Y. 2009.
Incongruence among mitochondrial, chloroplast and nuclear gene trees in Pinus
subgenus Strobus (Pinaceae). Journal of Plant Research 122(5), 509-521.
Van, L.M., Hintsteiner, W., Pötzelsberger, E., Schüler, S., Hasenauer, H. 2015. Intervarietal and intravarietal genetic structure in Douglas fir: nuclear SSRs bring novel insights into past population demographic processes,
phylogeography, and intervarietal hybridization. Ecology and Evolution 5(9), 1802-1817
Vendramin, G.G., Lelli, L., Rossi, P., Morgante, M. 1996. A set of primers
for the amplification of 20 chloroplast microsatellites in Pinaceae. Molecular
Ecology 5(4), 595-598.
Viard, F., El-Kassaby,
Y.A., Ritland, K. 2001. Diversity and genetic structure in populations of Pseudotsuga
menziesii (Pinaceae) at chloroplast microsatellite loci. Genome 44(3),
336-344.
Viveros-Viveros, H.,
Tapia-Olivares, B.L., Sáenz-Romero, C., Vargas-Hernández, J.J., López-Upton,
J., Santacruz-Varela, A., Ramírez-Valverde, G. 2010. Variación isoenzimática de
Pinus hartwegii Lindl. en un gradiente altitudinal en Michoacán, México.
Agrociencia 44(6), 723-733.
Wehenkel, C.,
Mariscal-Lucero, S.R., González-Elizondo, M.S., Aguirre-Galindo, V.A., Fladung,
M., López-Sánchez, C.A. 2020. Tall Pinus luzmariae
trees with genes from P. herrerae. PeerJ 26(8),
e86648.
Wei, X-X., Beaulieu, J., Khasa, D.P., Vargas-Hernández, J.V.,
López-Upton, J., Jaquish, B., Bousquet, J. 2011. Range-wide chloroplast and
mitochondrial DNA imprints reveal multiple lineages and complex biogeographic
history for Douglas-fir. Tree Genetics & Genomes 7, 1025-1040.
Whitney,
T.D., Gandhi, K.J., Hamrick, J.L., Lucardi, R.D. 2019. Extant population
genetic variation and structure of eastern white pine (Pinus strobus L.)
in the Southern Appalachians. Tree Genetics & Genomes 15,
74.
Xu, S., Tauer, C.G., Nelson, C.D. 2008. Genetic diversity within and
among populations of shortleaf pine (Pinus echinata Mill.) and loblolly
pine (Pinus taeda L.). Tree Genetics and Genomes 4(4), 859-868.
Yeh, F.C., Hu, X-S.
2005. Genetic structure and migration from mainland
to island populations in Abies procera Rehd. Genome 48(3), 461-473.
Yeh, F.C., Khalil, M.A.K., El-Kassaby, Y.A., Trust, D.C. 1986. Allozyme
variation in Picea mariana from Newfoundland: genetic diversity,
population structure, and analysis of differentiation. The Canadian Journal
of Forest Research 16(4), 713-720.
Zinck, J.W.R.,
Rajora, O.P. 2016. Post-glacial phylogeography and
evolution of a wide-ranging highly-exploited keystone forest tree, eastern
white pine (Pinus strobus) in North America: Single refugium, multiple
routes. BMC Evolutionary Biology 16, 56.
Anexo / Apendix
Tabla A1.
Valores estimados de diversidad y diferenciación genética entre poblaciones con
diferentes marcadores moleculares, para las especies de Abies de Norteamérica,
referencia bibliográfica de los estudios de los que se deriva la información y
su localización geográfica.
Table A1. Estimated values of diversity and genetic differentiation among populations with different molecular markers for Abies species from North America, bibliographic reference of the studies from which the information is derived and their geographic location.
P;
número de poblaciones, N; número de individuos, SSRs ADNcp; microsatélites de
cloroplasto, SSRs ADNn; microsatélites de núcleo, Ho; Heterocigosis observada,
He; Heterocigosis esperada.
P; number of populations, N; number of individuals,
SSRs ADNcp; chloroplast microsatellites, SSRs ADN; nuclear microsatellite, Ho;
observed heterozygosity, He; expected heterozygosity.
Tabla A2. Valores
estimados de diversidad y diferenciación genética entre poblaciones con
diferentes marcadores moleculares, para las especies de Picea de
Norteamérica, referencia bibliográfica de los estudios de los que se deriva la
información y su localización geográfica.
Table A2. Estimated values of diversity and genetic differentiation among populations with different molecular markers for Picea species from North America, bibliographic reference of the studies from which the information is derived and their geographic location.
P; número de poblaciones, N; número de individuos, SSRs
ADNcp; microsatélites de cloroplasto, SSRs ADNn; microsatélites nucleares, STS;
Sitio marcado con secuencia; SNPs; Polimorfismos de nucleótido único, Ho; Heterocigosis
observada, He; Heterocigosis esperada.
P; number of populations, N; number of individuals,
SSRs ADNcp; chloroplast microsatellites, SSRs ADN; nuclear microsatellite, STS;
Sequence-Tagged Sites; SNPs; Single Nucleotide Polymorphism, Ho; observed
heterozygosity, He; expected heterozygosity.
Tabla A3. Valores
estimados de diversidad y diferenciación genética entre poblaciones con
diferentes marcadores moleculares, para las especies de Pinus de
Norteamérica, referencia bibliográfica de los estudios de los que se deriva la
información y su localización geográfica.
Table A3. Estimated values of diversity and genetic differentiation among populations
with different molecular markers for Pinus species from North America,
bibliographic reference of the studies from which the information is derived
and their geographic location.
P;
número de poblaciones, N; número de individuos, SSRs ADNcp; microsatélites de
cloroplasto, SSRs ADNn; microsatélites nucleares, STS; Sitio marcado con
secuencia; SNPs; Polimorfismos de nucleótido único, Ho; Heterocigosis
observada, He; Heterocigosis esperada.
P; number of
populations, N; number of individuals, SSRs ADNcp; chloroplast microsatellites,
SSRs ADN; nuclear microsatellite, STS; Sequence-Tagged Sites; SNPs; Single
Nucleotide Polymorphism, Ho; observed heterozygosity, He; expected
heterozygosity.
Tabla A4. Valores
estimados de diversidad y diferenciación genética entre poblaciones con
diferentes marcadores moleculares, para las especies de Pseudotsuga de
Norteamérica, referencia bibliográfica de los estudios de los que se deriva la
información y su localización geográfica.
Table A4. Estimated values of diversity and genetic differentiation among populations
with different molecular markers for Pseudotsuga species from North
America, bibliographic reference of the studies from which the information is
derived and their geographic location.
P;
número de poblaciones, N; número de individuos, SSRs ADNcp; microsatélites de
cloroplasto, H; diversidad haplotípica, Ho; Heterocigosis observada, He;
Heterocigosis esperada.
P; number of
populations, N; number of individuals, SSRs ADNcp; chloroplast microsatellites,
H; haplotype diversity, Ho; observed heterozygosity, He; expected
heterozygosity.
Tabla A5. Lista
de especies incluidas en las distintas correlaciones de Pearson (r) de la Figura 2.
Table
A5. List of species included in the different
Pearson correlations (r) of Figure
2.
![]() , Alejandra
Moreno-Letelier2
, Alejandra
Moreno-Letelier2 ![]() , Patricia Delgado-Valerio3
, Patricia Delgado-Valerio3 ![]() , Daniel Piñero4
, Daniel Piñero4 ![]() , Carlos Ortiz-Martínez5
, Carlos Ortiz-Martínez5 ![]() , Alejandra
Vázquez-Lobo6,*
, Alejandra
Vázquez-Lobo6,* ![]()

